- Interazione ospite-specifica tra il rizobio e i partner vegetali
- Sviluppo di noduli determinato e indeterminato
- La crescita delle cellule simbiotiche comporta l’amplificazione del genoma ospite mediante cicli di endoreduplicazione
- Diversi destini di batteri che fissano l’azoto
- I peptidi ospiti governano la differenziazione dei batterioidi
- NCR247: Un esempio per l’effettuatore host multi-target
- Conclusione
- Dichiarazione sul conflitto di interessi
- Riconoscimenti
Interazione ospite-specifica tra il rizobio e i partner vegetali
I batteri che formano una simbiosi azotofissante con piante leguminose appartenenti a diversi gruppi di α-e β-proteobatteri sono collettivamente chiamati rhizobia (Chen et al., 2003; MacLean et al., 2007). Molti α-proteobatteri sono impegnati in interazioni a lungo termine con eucarioti più alti. Queste interazioni vanno dalla colonizzazione superficiale attraverso relazioni simbiotiche facoltative per obbligare gli stili di vita patogeni intracellulari o endosimbionti. I geni simbiotici necessari per la formazione di noduli, l’infezione delle cellule ospiti e la fissazione dell’azoto sono stati acquisiti dal trasferimento genico laterale che è la fonte primaria della diversità genetica di rhizobia. Pertanto, la rhizobia potrebbe essere più strettamente correlata agli agenti patogeni (come Agrobacterium o Brucella) che tra loro. Rhizobia tendono ad avere genomi di grandi dimensioni (fino a 10.5 Mbp) che in rapida crescita rhizobia sono dispersi su repliconi multipli (MacLean et al., 2007). Ad esempio, Sinorhizobium meliloti, l’endosimbionte delle specie Medicago, ha un genoma tripartito; un cromosoma 3.65 Mbp e due megaplasmidi, pSymA e pSymB (1.35 e 1.68 Mbp) entrambi indispensabili e portano la maggior parte dei geni simbiotici. Tuttavia, molti ceppi di S. meliloti contengono ulteriori plasmidi ausiliari di medie dimensioni e quindi, il genoma di S. meliloti può contenere fino a 9.000 geni (Barnett et al., 2001; Capela et al., 2001). A differenza della rhizobia, gli endosimbionti obbligati degli insetti di solito possiedono un genoma fortemente ridotto (160-450 Kbp) che garantisce la loro moltiplicazione e codici per alcune specifiche vie biosintetiche comprese quelle che soddisfano il bisogno dell’ospite (Moran et al., 2008; Price et al., 2011). Questi genomi incredibilmente ridotti sono tuttavia amplificati compensando il genoma diminuito con un contenuto di DNA poliploide.
I partner vegetali di rhizobia appartengono alla famiglia Leguminosae/Fabaceae. La simbiosi di fissaggio dell’azoto si è evoluta in diversi lignaggi, ma non tutti i legumi formano simbiosi. Finora sono note 12.000 specie di leguminose nodulate e ognuna ha il proprio partner Rhizobium. La simbiosi è innescata dalla fame di azoto della pianta ospite che deve selezionare il suo partner Rhizobium da miliardi di batteri nella rizosfera. Ciò è ottenuto dalla secrezione di molecole di segnale flavonoidi dalla radice che agiscono come chemio-attrattivi ma soprattutto come induttori dei geni di nodulazione del Rhizobium (Oldroyd, 2013). Questi geni sono necessari per la produzione di molecole di segnale batterico; i fattori Nod (NFs) che innescano il programma di sviluppo del nodulo nella pianta ospite (Walker e Downie, 2000). Le NFS sono molecole lipochitooligosaccharide che trasportano sostituzioni specifiche dell’ospite sui residui di zucchero terminali e sulle catene lipidiche caratteristiche, che sono riconosciute dai recettori ospiti di tipo LysM e sono necessarie sia per lo sviluppo di noduli che per l’infezione batterica. È interessante notare che l’antica simbiosi delle piante terrestri con funghi micorrizici arbuscolari (AM) opera con molecole di segnale lipochitooligosaccharide simili, i fattori Myc che sono percepiti da recettori di tipo LysM simili ma diversi come NFs (Abdel-Lateif et al., 2012; Oldroyd, 2013). I fattori Myc e NFs attivano una via di segnalazione comune ma dopo il coinvolgimento dei geni simbiotici comuni conservati nelle piante, i percorsi si discostano; uno che porta alla nodulazione, l’altro per la simbiosi AM.
L’infezione delle piante e la formazione di noduli sono processi intricati; I fattori di Nod svolgono ruoli distinti nell’organogenesi del nodulo e nell’infezione dei capelli della radice. Inoltre, oltre ai fattori Nod, vari polisaccaridi superficiali batterici sono cruciali per un’infezione efficiente (Fraysse et al., 2003). Nella maggior parte dei legumi, la rhizobia entra nell’ospite attraverso i peli della radice dove per invaginazione della membrana plasmatica si forma un filo di infezione (IT) che contiene i batteri moltiplicatori e cresce verso la corteccia della radice. Una modalità di infezione meno frequente e antica si verifica attraverso crepe sulla superficie della radice di alcuni legumi.
Sviluppo di noduli determinato e indeterminato
Lo sviluppo di noduli richiede la riattivazione mitotica delle cellule corticali che porta alla formazione di noduli primordiali che poi si differenziano in noduli radicali che fissano l’azoto fornendo condizioni microaerobiche nella zona centrale per il funzionamento dell’enzima nitrogenasi sensibile all’ossigeno nei batterioidi. A seconda della natura transitoria e persistente della proliferazione delle cellule ospiti, i noduli possono essere di tipo determinato o indeterminato (Terpolilli et al., 2012; Kondorosi et al., 2013). I noduli determinati non hanno meristema e contengono una popolazione omogenea di cellule simbiotiche. Noduli determinati si sviluppano ad esempio sulle radici di Phaseolus vulgaris e Lotus japonicus.
Al contrario, la divisione cellulare attiva viene mantenuta nei noduli indeterminati. Un meristema nodulare è presente nella regione apicale (zona I) che con la generazione costante di nuove cellule provoca una crescita continua e una forma allungata del nodulo. Le cellule che lasciano il meristema non si dividono più ed entrano in una fase di differenziazione. Il filo di infezione rilascia i batteri nelle cellule sommeristematiche, che si differenziano gradualmente lungo gli strati di cellule 12-15 della zona di infezione (zona II), portando allo sviluppo di cellule simbiotiche che fissano l’azoto nella zona del nodulo III (Figura 1; Franssen et al., 1992). Medicago sativa, M. truncatula, Vicia sativa e Pisum sativum sono esempi di piante che formano noduli indeterminati.
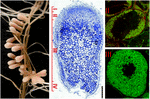
FIGURA 1. Struttura dei noduli radicali che fissano l’azoto formati nella simbiosi di S. meliloti-M. truncatula. Le diverse zone dei noduli sono indicate sulla sezione longitudinale del nodulo: (I) meristema, (II) zona di infezione, (III) zona di fissazione dell’azoto, (IV) zona di senescenza. Le cellule simbiotiche nella zona II contengono gli endosimbionti differenzianti mentre nella zona III il citoplasma ospite è completamente imballato con lunghi batterioidi che fissano l’azoto. Gli endosimbionti macchiati con Syto9 hanno fluorescenza verde.
La crescita delle cellule simbiotiche comporta l’amplificazione del genoma ospite mediante cicli di endoreduplicazione
L’allargamento estremo delle cellule vegetali può essere osservato sia nei noduli determinati che indeterminati. Il citoplasma di una cellula simbiotica che fissa l’azoto ospita circa 50.000 batterioidi. Per ospitare un numero così elevato di endosimbionti, le cellule ospiti crescono. Nei noduli di M. truncatula il volume delle cellule che fissano l’azoto è 80 volte più grande di quello delle cellule meristematiche diploidi. La crescita delle cellule infette avviene gradualmente nella zona II ed è la conseguenza della ripetuta endoreduplicazione (ER) del genoma senza mitosi. Nella zona II il macchinario del ciclo cellulare è ancora attivo, ma la mancanza di cicline mitotiche inibisce la mitosi e trasforma i cicli mitotici in cicli di endoreduplicazione (Cebolla et al., 1999). Ciò è ottenuto dalla proteina CCS52A dell’interruttore del ciclo cellulare che con la distruzione delle cicline mitotiche induce ripetuti cicli di duplicazione del genoma che portano alla formazione di cellule poliploidi a crescita graduale (Roudier et al., 2003; Kondorosi e Kondorosi, 2004). Nelle specie Medicago i livelli di ploidy possono raggiungere 64C che rappresentano un contenuto di DNA 64 volte superiore rispetto alle cellule aploidi (C corrisponde al contenuto di DNA aploide; Vinardell et al., 2003). La down-regolazione di CCS52A in M. truncatula non ha avuto alcun effetto sulla formazione del primordio, ma è stata dannosa per la differenziazione dei noduli, indicando che i cicli ER e la formazione di grandi cellule altamente poliploidi sono essenziali per il funzionamento dei noduli (Vinardell et al., 2003). È interessante notare che le cellule corticali contenenti funghi AM sono anche poliploidi, così come le cellule radicali giganti che alimentano i nematodi (Favery et al., 2002; Genere et al., 2008). Allo stesso modo, le cellule simbiotiche degli insetti, i batteriociti che ospitano endosimbionti intracellulari sono anche grandi e poliploidi (Nakabachi et al., 2010). Nelle piante angiosperme, la poliploidia è frequente e lo specifico modello ereditario della poliploidia in diversi organi, tessuti e tipi di cellule suggerisce che potrebbe essere una fonte importante della fisiologia specializzata delle cellule ospiti (Nagl, 1976; Edgar et al., 2014). Oltre alla crescita cellulare, le copie multiple del gene, la mancanza di condensazione cromosomica può contribuire ad attività trascrizionali e metaboliche più elevate. Tuttavia, l’associazione della poliploidia con diverse funzioni cellulari suggerisce un impatto della poliploidia anche sull’architettura dei nucleosomi e sull’epigenoma che controlla l’attivazione o la repressione di specifiche regioni genomiche. Di conseguenza, il contenuto del genoma poliploide delle cellule simbiotiche sembra essere un prerequisito per la differenziazione dei noduli e per l’espressione della maggior parte dei geni ospiti simbiotici (Maunoury et al., 2010).
Diversi destini di batteri che fissano l’azoto
I batteri rilasciati dall’IT sono presenti nel citoplasma ospite come strutture organelliche, chiamate simbiosomi. I batteri non hanno alcun contatto diretto con il citoplasma in quanto sono circondati da una membrana peribacteroide, nota anche come membrana simbiosoma (SM). Il bacteroide, l’SM e lo spazio tra di loro comprendono il simbiosoma (Catalano et al., 2004). L’SM durante la sua formazione riflette la sua origine della membrana plasmatica, le successive modifiche della sua composizione aprono nuovi ruoli specializzati nell’interfaccia host-endosimbiont (Limpens et al., 2009; Ivanov et al., 2012; Brear et al., 2013; Sinharoy et al., 2013). I bacteroids si moltiplicano nelle cellule crescenti del nodulo ospite ad una determinata densità delle cellule, si adattano allo stile di vita endosymbiotic ed alle circostanze microaerobic e maturano ai bacteroids azoto-fissanti. La forma e la fisiologia dei batterioidi possono essere, tuttavia, sorprendentemente diverse nei vari legumi. In alcuni ospiti di legumi, i bacteroidi che fissano l’azoto hanno la stessa morfologia delle cellule coltivate; questo tipo di bacteroidi può tornare alla forma di vita libera. In altre associazioni, i batterioidi sono irreversibilmente trasformati in endosimbionti poliploidi, ingrossati, non coltivabili. Questi batterioidi terminali differenziati possono essere allungati e persino ramificati e da 5 a 10 volte più lunghi delle cellule viventi libere o possono essere sferici da 8 ad almeno 20 volte il genoma amplificato a seconda dell’ospite (Mergaert et al., 2006; Nakabachi et al., 2010). La differenziazione terminale dei batterioidi è controllata dall’ospite, evoluta in più rami della famiglia delle leguminose, indicando il vantaggio dell’ospite e probabilmente prestazioni simbiotiche più elevate (Oono et al., 2010). La differenziazione batteroide terminale è la migliore chiarita nella simbiosi di S. meliloti-M. truncatula. In M. noduli truncatula, gli eventi più visibili di differenziazione batterioide terminale si verificano nella zona II. Moltiplicazione dei batterioidi si ferma nel mezzo della zona II dove l’allungamento cellulare e l’amplificazione uniforme dei repliconi multipli da endoreduplication cicli iniziano. Lungo 2-3 strati cellulari al confine della zona II e III (chiamato interzone) la crescita improvvisa di batterioidi è visibile raggiungendo praticamente la loro dimensione finale, tuttavia, la fissazione dell’azoto avviene solo nella zona III.
I peptidi ospiti governano la differenziazione dei batterioidi
Il confronto dei trascrittomi nodulari dei legumi con differenziazione dei batterioidi reversibile e irreversibile ha rivelato l’esistenza di diverse centinaia di piccoli geni che erano presenti solo nel genoma di quelle piante ospiti in cui la differenziazione dei batterioidi era terminale. In M. truncatula le cellule nodulari producono almeno 600 peptidi simbiotici noduli-specifici (symPEPs). I geni symPEP sono attivati solo nelle cellule simbiotiche poliploidi infette da S. meliloti (Kevei et al., 2002; Mergaert et al., 2003), tuttavia alcuni set in precedenza, altri durante le fasi successive dello sviluppo del nodulo. Una grande porzione, più di 500 geni codificano peptidi ricchi di cisteina (NCR) nodulo-specifici (Mergaert et al., 2003; Alunni et al., 2007; Nallu et al., 2014). I peptidi NCR sono mirati ai batterioidi e quando la loro consegna agli endosimbionti è stata bloccata, la differenziazione dei batterioidi è stata abolita dimostrando che i peptidi sono responsabili della differenziazione terminale dei batterioidi di S. meliloti (Van de Velde et al., 2010). La varietà ad alta sequenza e i modelli di espressione caratteristica dei geni NCR suggeriscono la diversità nelle loro funzioni, modalità di azione e bersagli batterici a diversi stadi di maturazione dei batteri (Figura 2). Tuttavia, perché la cellula ospite produce un arsenale di NCRS? Quale può essere il vantaggio di un repertorio peptidico così diversificato? È necessario per l’interazione dell’ospite con vari batteri? I partner simbiotici di M. truncatula sono S. meliloti e S. medicae, tuttavia nel terreno ci sono innumerevoli varianti di ceppo di entrambe le specie. M. truncatula è anche rappresentato da molti diversi ecotipi e adesioni che differiscono nel numero, nelle sequenze e nel profilo di espressione dei geni NCR e nelle loro interazioni simbiotiche con diversi ceppi di S. meliloti e S. medicae (Nallu et al., 2014; Roux et al., 2014). Mentre un nodulo contiene un singolo tipo di batterio, i diversi noduli sullo stesso apparato radicale possono possedere popolazioni batteriche distinte. È possibile che la pianta che riconosce i vari endosimbionti li manipoli con un repertorio ceppo-specifico di peptidi. Queste differenze possono aggiungere un ulteriore livello di controllo per la specificità ospite-simbionte e quindi per l’efficienza della nodulazione.

FIGURA 2. Espressione differenziale dei geni symPEP nei noduli di M. truncatula. Segnale nero: ibridazione in situ, segnale blu: attività GUS del promotore symPEP-fusioni GUS in noduli transgenici.
Sebbene i symPEPs rappresentino classi peptidiche uniche, le loro strutture assomigliano ai peptidi antimicrobici (AMPs). Gli AMPLIFICATORI con ampio spettro di attività microbica di uccisione delle cellule sono più frequentemente cationici provocando la morte cellulare per formazione di pori, interruzione della membrana e conseguente lisi delle cellule microbiche. Il fatto che la capacità di divisione cellulare sia definitivamente persa durante la differenziazione endosimbionta indica che almeno alcuni symPEPs hanno attività antimicrobiche. Il trattamento dei batteri con NCRS cationici sintetici ha infatti provocato una rapida ed efficiente eliminazione dose-dipendente di vari batteri Gram-negativi e Gram-positivi, inclusi importanti patogeni umani e vegetali (Van de Velde et al., 2010; Tiricz et al., 2013). Questo effetto di ex-planta killing correlato alla permeabilizzazione delle membrane microbiche, tuttavia, i symPEPs nel loro ambiente naturale – nelle cellule dei noduli-non permeabilizzano le membrane batteriche e non uccidono gli endosimbionti. Molto probabilmente le concentrazioni di peptide nei noduli sono significativamente inferiori a quelle applicate nei test in vitro. Inoltre i peptidi cationici sono prodotti insieme ai peptidi anionici e neutri nella stessa cellula e la possibile combinazione di poche decine o centinaia di peptidi con varie cariche e idrofobicità potrebbe neutralizzare l’effetto battericida diretto dei peptidi cationici.
Il coinvolgimento di AMP o peptidi simili ad AMP non è unico per la simbiosi Rhizobium-legume. Nel punteruolo sitofilo, le cellule simbiotiche producono il peptide antimicrobico coleoptericina – A (ColA) che provoca lo sviluppo di endosimbionti filamentosi giganti inibendo la divisione cellulare e protegge i tessuti degli insetti vicini dall’invasione batterica (Login et al., 2011). In questo sistema un singolo peptide è sufficiente per la differenziazione dell’endosimbionte obbligato trasmesso verticalmente a differenza dei noduli che operano con centinaia di symPEPs e possono ospitare innumerevoli varianti di ceppo come loro endosimbionti. Nella simbiosi afide-Buchnera, le cellule ospiti producono anche peptidi specifici per batteriociti inclusi peptidi ricchi di cisteina (BCR) che assomigliano ai peptidi Medicago NCR, tuttavia le funzioni di questi peptidi simbiotici non sono state ancora riportate (Shigenobu e Stern, 2013).
NCR247: Un esempio per l’effettuatore host multi-target
Analisi del trascrittoma di M. noduli truncatula in diverse fasi del loro sviluppo, microdissezione laser delle regioni nodulari, ibridazione in situ, immunolocalizzazione di peptidi selezionati e fusioni di geni promotore-reporter symPEP in noduli transgenici consentono di mappare l’azione dei singoli peptidi nelle cellule simbiotiche dall’infezione precoce fino allo stato di fissazione tardiva dell’azoto. NCR247 è espresso negli strati cellulari più vecchi della zona II e nell’interzone dove la divisione cellulare batterica si ferma e si verifica un notevole allungamento degli endosimbionti (Farkas et al., 2014). Questo piccolo peptide cationico ha efficacemente ucciso vari microbi in vitro e l’analisi in silico ha indicato le sue estreme capacità di legame proteico. NCR247 marcato con FITC è entrato nel citosol batterico dove le sue interazioni con numerose proteine batteriche erano possibili. I partner vincolanti sono stati identificati mediante trattamento di batteri o batteri di S. meliloti con peptidi StrepII/FLAG-tagged seguiti da cromatografia di affinità e identificazione di partner interagenti con LC-MS / MS e analisi occidentale (Farkas et al., 2014).
Uno degli interattori era la proteina di divisione cellulare FtsZ che svolgeva un ruolo primario cruciale nella divisione cellulare. Un certo numero di peptidi antibiotici è noto per esercitare un effetto battericida o batteriostatico attraverso l’interazione con FtsZ, inibendo la sua polimerizzazione ostacolando così la corretta formazione di Z-ring e setto (Handler et al., 2008). NCR247 è stato co-purificato con FtsZ dal citoplasma batterico ed è stato dimostrato di interrompere la formazione del setto. NCR035 esibendo in vitro anche l’effetto battericida e prodotto nelle stesse cellule simbiotiche come NCR247 si accumula al setto di divisione che indica l’azione simultanea o consecutiva di questi peptidi e l’evoluzione delle strategie multiple dell’ospite per inibire la proliferazione di endosymbiont. Un altro studio ha dimostrato che l’espressione di importanti geni di divisione cellulare, inclusi i geni necessari per la funzione dell’anello Z, erano fortemente attenuati nelle cellule trattate da NCR247 (Penterman et al., 2014). Il pretrattamento di batteri con concentrazioni sub-letali di NCR247 ha abolito la localizzazione di FITC-NCR035 al setto e ha provocato l’allungamento cellulare (Farkas et al., 2014).
Le proteine ribosomiali erano i partner interagenti NCR247 più abbondanti. NCR247 è stato osservato per inibire fortemente la sintesi proteica batterica in modo dose-dipendente sia in vivo che in vitro (Farkas et al., 2014). Questi risultati hanno suggerito che una modalità dell’azione peptidica NCR247 si lega ai ribosomi sia nelle cellule batteriche che nei batterioidi. È interessante notare che nei batterioidi sono stati osservati un modello alterato e una ridotta complessità delle proteine interagenti. Di conseguenza il livello generale di espressione delle proteine ribosomiali era in media 20 volte inferiore nei batteroidi rispetto alle cellule viventi libere con diversa abbondanza relativa di trascritti delle singole proteine ribosomiali. La diversificazione dei ribosomi nei batterioidi può avere un ruolo significativo contribuendo alla traduzione avanzata di proteine specifiche, supportando così la fisiologia specializzata e energivora della funzione di fissazione dell’azoto altamente abbondante.
Lo chaperon GroEL era anche un partner interagente diretto di NCR247 (Farkas et al., 2014). Delle 5 proteine di GroEL, GroEL1 o GroEL2 è sufficiente per la sopravvivenza mentre GroEL1 espresso ad alto livello nel nodulo è essenziale per la simbiosi (Bittner et al., 2007). È necessario per l’attivazione completa dei geni di nodulazione e l’assemblaggio del complesso nitrogenasi. GroEL possiede un’estrema versatilità funzionale interagendo con centinaia di proteine. L’interazione NCR247-GroEL1 può avere conseguenze direttamente su GroEL ma indirettamente anche sui substrati di GroEL e sui processi biologici associati. L’assenza di GroEL1 ha influenzato gravemente l’infezione batterica e il mantenimento e la differenziazione dei batterioidi che dimostrano una necessità generale di GroEL1 in tutte le fasi dello sviluppo del nodulo di fissaggio dell’azoto.
Il coinvolgimento di Groel e peptidi ospiti nelle interazioni microbo-ospite non è unico per la simbiosi Rhizobium-legume. Nelle cellule simbiotiche del punteruolo la coleottericina-A (ColA) interagisce anche con GroEL (Login et al., 2011). GroEL svolge anche un ruolo importante nel mantenimento degli endosimbionti (Moran, 1996; Kupper et al., 2014). Poiché la maggior parte dei sistemi simbiotici sono ancora inesplorati e gli strumenti genomici e proteomici ad alto rendimento sono disponibili solo di recente, possiamo solo prevedere che la differenziazione endosimbionta mediata dai peptidi ospiti, allo stesso modo l’amplificazione del genoma delle cellule ospiti e gli endosimbionti terminali differenziati sono strategie generali di simbiosi.
Conclusione
I batteri simbiotici e patogeni usano approcci simili per interagire con i loro ospiti e sopravvivere all’interno delle cellule ospiti, anche se i risultati di queste interazioni sono sorprendentemente diversi. Piante e animali possono generare risposte immunitarie innate ai microrganismi sulla percezione di MAMPs (modelli molecolari associati a microrganismi). Questa percezione si traduce nell’attivazione di cascate di segnalazione e nella produzione di effettori antimicrobici. Peptidi ospite AMP-like come il M. i peptidi di truncatula NCR o il peptide antimicrobico del punteruolo ColA svolgono ruoli chiave e sfaccettati nel controllo della moltiplicazione e della differenziazione degli endosimbionti, limitando così la presenza di batteri alle cellule simbiotiche. Pertanto, gli organismi ospiti utilizzano questi peptidi effettori per domare e persino assumere invasori microbici selezionati per il servizio.
Dichiarazione sul conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Riconoscimenti
Il lavoro nei nostri laboratori è supportato dalla sovvenzione avanzata”SYM-BIOTICS” del Consiglio Europeo della ricerca a Éva Kondorosi (sovvenzione numero 269067) e da TÁMOP-4.2.2.A-11/1 / KONV-2012-0035 sostenuto dall’Unione Europea e cofinanziato dal Fondo Sociale Europeo.il suo nome deriva dal nome della città, che si trova nella parte sud-occidentale del paese. Il ruolo dei flavonoidi nella creazione di endosymbioses radici delle piante con funghi micorriza arbuscular, rhizobia e batteri Frankia. Segnale impianto. Comportati bene. 7, 636–641. doi: 10.4161 / psb.20039
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Alunni, B., Kevei, Z., Redondo-Nieto, M., Kondorosi, A., Mergaert, P., and Kondorosi, E. (2007). Genomic organization and evolutionary insights on GRP and NCR genes, two large nodule-specific gene families in Medicago truncatula. Mol. Plant Microbe Interact. 20, 1138–1148. doi: 10.1094/MPMI-20-9-1138
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Barnett, M. J., Fisher, R. F., Jones, T., Komp, C., Abola, A. P., Barloy-Hubler, F., et al. (2001). Sequenza nucleotidica e funzioni previste dell’intero Sinorhizobium meliloti pSymA megaplasmid. Proc. Natl. Acad. Sic. USA 98, 9883-9888. doi: 10.1073 / pnas.161294798
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Bittner, A. N., Foltz, A., and Oke, V. (2007). Solo uno dei cinque geni groEL è richiesto per la vitalità e la simbiosi di successo in Sinorhizobium meliloti. J. Batteriolo. 189, 1884–1889. doi: 10.1128 / JB.01542-06
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Brear, E. M., Day, D. A., and Smith, P. M. (2013). Iron: an essential micronutrient for the legume-Rhizobium symbiosis. Front. Plant Sci. 4:359. doi: 10.3389/fpls.2013.00359
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Catalano, C. M., Lane, W. S., and Sherrier, D. J. (2004). Biochemical characterization of symbiosome membrane proteins from Medicago truncatula root nodules. Electrophoresis 25, 519–531. doi: 10.1002/elps.200305711
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Capela, D., Barloy-Hubler, F., Gouzy, J., Bothe, G., Ampe, F., Batut, J.,et al. (2001). Analisi della sequenza cromosomica del legume simbionte Sinorhizobium meliloti ceppo 1021. Proc. Natl. Acad. Sic. USA 98, 9877-9882. doi: 10.1073 / pnas.161294398
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Cebolla, A., Vinardell, J. M., Kiss, E., Oláh, B., Roudier, F., Kondorosi, A.,et al. (1999). L’inibitore mitotico ccs52 è necessario per l’endoreduplicazione e l’allargamento delle cellule dipendente dalla ploidia nelle piante. EMBO J. 18, 4476-4484. doi: 10.1093/emboj/18.16.4476
CrossRef Full Text
Chen, WM, Moulin, L., Bontemps, C., Vandamme, P., Béna, G., and Boivin-Masson, C. (2003). La fissazione simbiotica dell’azoto del legume da parte dei β-proteobatteri è molto diffusa in natura. J. Batteriolo. 185, 7266–7272. doi: 10.1128 / JB.185.24.7266-7272.2003
CrossRef Full Text
Edgar, BA, Zielke, N., and Gutierrez, C. (2014). Endocicli: un’innovazione evolutiva ricorrente per la crescita cellulare post-mitotica. NAT. Rev. Mol. Cellula. Biol. 15, 197–210. doi: 10.1038 | nrm3756
Pubmed Abstract/Pubmed Full Text/CrossRef Full Text
Farkas, A., Maróti, G., Dürgõ, H., Györgypál, Z., Lima, R. M., Medzihradszky, K. F.,et al. (2014). Il peptide simbiotico Medicago truncatula NCR247 contribuisce alla differenziazione dei batteri attraverso meccanismi multipli. Proc. Natl. Acad. Sic. U. S. A. 111, 5183-5188. doi: 10.1073 / pnas.1404169111
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Favery, B., Complainville, A., Vinardell, J. M., Lecomte, P., Vaubert, D., Mergaert, P.,et al. (2002). The endosymbiosis-induced genes ENOD40 and CCS52a are involved in endoparasitic-nematode interactions in Medicago truncatula. Mol. Plant-Microbe Interact. 15, 1008–1013. doi: 10.1094/MPMI.2002.15.10.1008
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Franssen, H. J., Vijn, I., Yang, W. C., and Bisseling, T. (1992). Developmental aspects of the Rhizobium-legume symbiosis. Plant Mol. Biol. 19, 89–107. doi: 10.1007
CrossRef Full Text
Fraysse, N., Couderc, F., and Poinsot, V. (2003). Surface polysaccharide involvement in establishing the Rhizobium–legume symbiosis. Eur. J. Biochem. 270, 1365–1380. doi: 10.1046/j.1432-1033.2003.03492.x
CrossRef Full Text
Genre, A., Chabaud, M., Faccio, A., Barker, D. G., and Bonfante, P. (2008). L’assemblea dell’apparato di prepenetrazione precede e predice i modelli di colonizzazione dei funghi micorrizici arbuscular all’interno della corteccia della radice sia di Medicago truncatula che di Daucus carota. Cellula vegetale 20, 1407-1420. doi: 10.1105 / tpc.108.059014
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Gestore, A. A., Lim, J. E., e Losick, R. (2008). Inibitore peptidico della citochinesi durante la sporulazione in Bacillus subtilis. Mol. Microbiolo. 68, 588–599. doi: 10.1111 / j. 1365-2958. 2008.06173.x
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Ivanov, S., Fedorova, E. E., Limpens, E., De Mita, S., Genre, A., Bonfante, P.,et al. (2012). Rhizobium–legume symbiosis shares an exocytotic pathway required for arbuscule formation. Proc. Natl. Acad. Sci. U.S.A. 109, 8316–8321. doi: 10.1073/pnas.1200407109
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Kevei, Z., Vinardell, J. M., Kiss, G. B., Kondorosi, A., and Kondorosi, E. (2002). Le proteine ricche di glicina codificate da una famiglia genica nodulare specifica sono implicate in diverse fasi dello sviluppo simbiotico del nodulo in Medicago spp. Mol. Il microbo vegetale interagisce. 15, 922–931. doi: 10.1094 / MPMI.2002.15.9.922
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Kondorosi, E., and Kondorosi, A. (2004). Endoreduplicazione e attivazione del complesso di promozione delle anafasi durante lo sviluppo delle cellule simbiotiche. FEBS Lett. 567, 152–157. doi: 10.1016 / j. febslet.2004.04.075
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Kondorosi, E., Mergaert, P., and Kereszt, A. (2013). Un paradigma per la vita endosimbiotica: differenziazione cellulare dei batteri Rhizobium provocata da fattori vegetali ospiti. Annu. Rev. Microbiol. 67, 611–628. doi: 10.1146/annurev-micro-092412-155630
CrossRef Full Text
Kupper, M., Gupta, S. K., Feldhaar, H., e Gross, R. (2014). Ruoli versatili del chaperonin GroEL nelle interazioni microrganismo-insetto. FEMS Microbiol. Lett. 353, 1–10. doi: 10.1111 | 1574-6968.12390
Pubmed Abstract/Pubmed Full Text/CrossRef Full Text
Limpens, E., Ivanov, S., van Esse, W., Voets, G., Fedorova, E., and Bisseling, T. (2009). I simbiosomi Medicago N2-fixing acquisiscono il marcatore di identità endocitica Rab7 ma ritardano l’acquisizione dell’identità vacuolare. Cellula vegetale 21, 2811-2828. doi: 10.1105 / tpc.108.064410
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Login, F. H., Balmand, S., Vallier, A., Vincent-Monégat, C., Vigneron, A., Weiss-Gayet, M., et al. (2011). I peptidi antimicrobici tengono sotto controllo gli endosimbionti degli insetti. Scienza 334, 362-365. doi: 10.1126 / scienza.1209728
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
MacLean, A. M., Finan, T. M., and Sadowsky, M. J. (2007). Genomi dei batteri simbiotici che fissano l’azoto dei legumi. Impianto Physiol. 144, 615–622. doi: 10.1104/pp. 107.101634
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Maunoury, N., Redondo-Nieto, M., Bourcy, M., Van de Velde, W., Alunni, B., Laporte, P., et al. (2010). La differenziazione delle cellule simbiotiche e degli endosimbionti è accoppiata a due interruttori del trascrittoma nella nodulazione di Medicago truncatula. PLoS UNO 5: e9519. doi: 10.1371 / giornale.pone.0009519
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Mergaert, P., Nikovics, K., Kelemen, Z., Maunoury, N., Vaubert, D., Kondorosi, A.,et al. (2003). Una nuova famiglia a Medicago truncatula composta da più di 300 geni noduli specifici che codificano per piccoli polipeptidi secreti con motivi di cisteina conservati. Impianto Physiol. 132, 161–173. doi: 10.1104/pp.102.018192
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Mergaert, P., Uchiumi, T., Alunni, B., Evanno, G., Cheron, A., Catrice, O.,et al. (2006). Eukaryotic control on bacterial cell cycle and differentiation in the Rhizobium-legume symbiosis. Proc. Natl. Acad. Sci. U.S.A. 103, 5230–5235. doi: 10.1073/pnas.0600912103
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Moran, N. A. (1996). Evoluzione accelerata e cricchetto di Muller nei batteri endosimbiotici. Proc. Natl. Acad. Sic. USA 93, 2873-2878. doi: 10.1073 / pnas.Per la prima volta, il programma è stato pubblicato nel 2008, mentre per la seconda nel 2008 è stato pubblicato nel 2008. Genomica ed evoluzione dei simbionti batterici ereditari. Annu. Rev. Genet. 42, 165–690. doi: 10.1146 / annurev.genet.Il suo nome deriva dal nome della città di Nakabachi, A., Koshikawa, S., Miura, T., e Miyagishima, S. (2010). Dimensione del genoma di Pachypsylla venusta (Hemiptera: Psyllidae) e la ploidia del suo bacteriocyte, la cellula ospite simbiotica che ospita batteri mutualistici intracellulari con il genoma cellulare più piccolo. Toro. Entomolo. Res. 100, 27-33. doi: 10.1017 / S0007485309006737
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nagl, W. (1976). Endoreduplicazione del DNA e politenia intese come strategie evolutive. Natura 261, 614-615. doi: 10.1038/261614a0
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Nallu, S., Silverstein, K. A., Zhou, P., Young, ND, e Vandenbosch, KA (2014). Modelli di divergenza di una grande famiglia di peptidi ricchi di cisteina nodulare in adesioni di Medicago truncatula. Impianto J. 78, 697-705. doi: 10.1111 / tpj.12506
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Oldroyd, GE (2013). Parla, amico ed entra: sistemi di segnalazione che promuovono associazioni simbiotiche benefiche nelle piante. NAT. Rev. Microbiol. 11, 252–263. doi: 10.1038/nrmicro2990
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Oono, R., Schmitt, I., Sprent, J. I., and Denison, R. F. (2010). Multiple evolutionary origins of legume traits leading to extreme rhizobial differentiation. New Phytol. 187, 508–520. doi: 10.1111/j.1469-8137.2010.03261.x
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Penterman, J., Abo, R. P., De Nisco, N. J., Arnold, M. F., Longhi, R., Zanda, M.,et al. (2014). I peptidi della pianta ospite suscitano una risposta trascrizionale per controllare il ciclo cellulare di Sinorhizobium meliloti durante la simbiosi. Proc. Natl. Acad. Sic. U. S. A. 111, 3561-3566. doi: 10.1073 / pnas.1400450111
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Price, D. R., Duncan, R. P., Shigenobu, S., and Wilson, AC (2011). Espansione del genoma ed espressione differenziale dei trasportatori di aminoacidi all’interfaccia simbiotica afide / buchnera. Mol. Biol. Evol. 28, 3113–3126. doi: 10.1093/molbev/msr140
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Roudier, F., Fedorova, E., Lebris, M., Lecomte, P., Györgyey, J., Abad, P.,et al. (2003). The Medicago species A2-type cyclin is auxin regulated and involved in meristem formation but dispensable for endoreduplication-associated developmental programs. Plant Physiol. 131, 1091–1103. doi: 10.1104/pp.102.011122
CrossRef Full Text
Roux, B., Rodde, N., Jardinaud, M. F., Timmers, T., Sauviac, L., Cottret, L.,et al. (2014). Un’analisi integrata dell’espressione genica vegetale e batterica in noduli di radice simbiotica utilizzando microdissezione a cattura laser accoppiata al sequenziamento dell’RNA. Impianto J. 77, 817-837. doi: 10.1111 / tpj.12442
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Shigenobu, S., and Stern, DL (2013). Gli afidi hanno evoluto nuove proteine secrete per simbiosi con endosimbionte batterico. Proc. Biol. Sic. 280, 20121952. doi: 10.1098 / rspb.2012.1952
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Sinharoy, S., Torres-Jerez, I., Bandyopadhyay, K., Kereszt, A., Pislariu, CI, Nakashima, J.,et al. (2013). Il regolatore del fattore di trascrizione C2H2 della differenziazione del simbiosoma reprime la trascrizione del gene VAMP721a della via secretoria e promuove lo sviluppo del simbiosoma in Medicago truncatula. Cellula vegetale 25, 3584-3601. doi: 10.1105 / tpc.113.114017
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Terpolilli, J. J., Hood, G. A., and Poole, P. S. (2012). What determines the efficiency of N(2)-fixing Rhizobium-legume symbioses? Adv. Microb. Physiol. 60, 325–389. doi: 10.1016/B978-0-12-398264-3.00005-X
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Tiricz, H., Szûcs, A., Farkas, A., Pap, B., Lima, R. M., Maróti, G.,et al. (2013). Antimicrobial nodule-specific cysteine-rich peptides induce membrane depolarization associated changes in Sinorhizobium meliloti. Appl. Env. Microbiol. 79, 6737–6746. doi: 10.1128/AEM.01791-13
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Van de Velde, W., Zehirov, G., Szatmari, A., Debreczeny, M., Ishihara, H., Kevei, Z.,et al. (2010). Plant peptides govern terminal differentiation of bacteria in symbiosis. Science 327, 1122–1126. doi: 10.1126/science.1184057
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Vinardell, J. M., Fedorova, E., Cebolla, A., Kevei, Z., Horvath, G., Kelemen, Z.,et al. (2003). L’endoreduplicazione mediata dall’attivatore complesso che promuove l’anafase CCS52A è necessaria per la differenziazione cellulare simbiotica nei noduli di Medicago truncatula. Cellula vegetale 15, 2093-2105. doi: 10.1105 / tpc.014373
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Walker, SA, e Downie, JA (2000). Ingresso di Rhizobium leguminosarum bv. viciae nei peli della radice richiede una specificità minima del fattore Nod, ma la successiva crescita del filo di infezione richiede nodO o nodO. Mol. Pianta-Microbo Interagire.13, 54–62. doi: 10.1094 / MPMI.2000.13.7.754
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text