In quasi tutte le proteine Fe–S, i centri Fe sono tetraedrici ei ligandi terminali sono centri di zolfo tiolato da residui di cisteinile. I gruppi solfuro sono due o tre coordinati. Tre tipi distinti di cluster Fe–S con queste caratteristiche sono più comuni.
2Fe–2S clustersEdit

Il più semplice polimetallici sistema, il cluster è costituito da due ioni di ferro collegato da due ioni solfuri e coordinato da quattro cysteinyl ligandi (in Fe2S2 ferredoxins) o da due cisteine e due histidines (in Rieske proteine). Le proteine ossidate contengono due ioni Fe3+, mentre le proteine ridotte contengono uno Fe3 + e uno Fe2+ ion. Queste specie esistono in due stati di ossidazione, (FeIII)2 e FeIIIFeII. CDGSH iron sulfur domain è anche associato a cluster 2Fe-2S.
4FE–4S clustersEdit
Un motivo comune presenta quattro ioni di ferro e quattro ioni solfuro posti ai vertici di un cluster di tipo cubano. I centri Fe sono in genere ulteriormente coordinati da leganti cisteinilici. Le proteine a trasferimento elettronico (ferredossine) possono essere ulteriormente suddivise in ferredossine a basso potenziale (di tipo batterico) e ad alto potenziale (HiPIP). Le ferredossine a basso e alto potenziale sono correlate dal seguente schema redox:
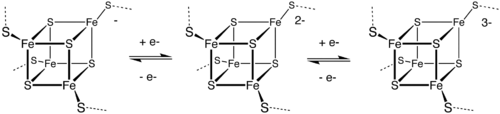
In HiPIP, il cluster navette tra (Fe4S42+) e (Fe4S43+). I potenziali per questa coppia redox vanno da 0,4 a 0,1 V. Nelle ferredossine batteriche, la coppia di stati di ossidazione è (Fe4S4+) e (Fe4S42+). I potenziali per questa coppia redox vanno da -0,3 a -0,7 V. Le due famiglie di cluster 4Fe-4S condividono lo stato di ossidazione Fe4S42+. La differenza nelle coppie redox è attribuita al grado di legame dell’idrogeno, che modifica fortemente la basicità dei ligandi cisteinil tiolato. Un’ulteriore coppia redox, che è ancora più riducente delle ferredossine batteriche, è implicata nella nitrogenasi.
Alcuni cluster 4Fe–4S legano i substrati e sono quindi classificati come cofattori enzimatici. Nell’aconitasi, il cluster Fe–S lega l’aconitato all’unico centro Fe privo di un ligando tiolato. Il cluster non subisce redox, ma serve come catalizzatore acido di Lewis per convertire il citrato in isocitrato. Negli enzimi radicali SAM, il cluster si lega e riduce la S-adenosilmetionina per generare un radicale, che è coinvolto in molte biosintesi.
3FE–4S clustersEdit
Le proteine sono anche note per contenere centri, che presentano un ferro in meno rispetto ai nuclei più comuni. Tre ioni solfuro ponte due ioni di ferro ciascuno, mentre il quarto solfuro ponti tre ioni di ferro. I loro stati di ossidazione formale possono variare da+ (all-Fe3 + form) a 2 – (all-Fe2+ form). In un certo numero di proteine ferro–zolfo, il cluster può essere convertito reversibilmente dall’ossidazione e dalla perdita di uno ion ferro in un cluster. Ad esempio, la forma inattiva di aconitase possiede un ed è attivata da aggiunta di Fe2 + e reductant.
Altri clustersEdit Fe–S
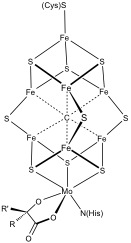
Sistemi polimetallici più complessi sono comuni. Gli esempi includono sia i cluster 8Fe che 7Fe in nitrogenasi. Il monossido di carbonio deidrogenasi e la-idrogenasi presentano anche insoliti cluster Fe-S. Uno speciale cluster coordinato a 6 cisteina è stato trovato nelle idrogenasi legate alla membrana tolleranti all’ossigeno.