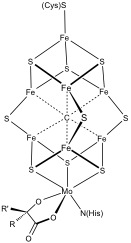
In fast allen Fe–S-Proteinen sind die Fe-Zentren tetraedrisch und die terminalen Liganden sind Thiol-Schwefel-Zentren aus Cysteinylresten. Die Sulfidgruppen sind entweder zwei- oder dreifach koordiniert. Drei verschiedene Arten von Fe-S-Clustern mit diesen Merkmalen sind am häufigsten.
2FE–2S–Clusterbearbeiten

Das einfachste polymetallische System, der Cluster, besteht aus zwei Eisenionen, die von zwei Sulfidionen überbrückt und von vier Cysteinylliganden (in Fe2S2-Ferredoxinen) oder durch zwei Cysteine und zwei Histidine (in Rieske-Proteinen). Die oxidierten Proteine enthalten zwei Fe3+ -Ionen, während die reduzierten Proteine ein Fe3 + – und ein Fe2+ -Ion enthalten. Diese Arten existieren in zwei Oxidationsstufen, (FeIII) 2 und FeIIIFeII. Die CDGSH-Eisen-Schwefel-Domäne ist auch mit 2Fe-2S-Clustern assoziiert.
4Fe–4S clusterbearbeiten
Ein gemeinsames Motiv besteht aus vier Eisenionen und vier Sulfidionen, die an den Eckpunkten eines Cuban-Clusters angeordnet sind. Die Fe-Zentren werden typischerweise durch Cysteinylliganden weiter koordiniert. Die Elektronentransferproteine (Ferredoxine) können weiter in niedrigpotentielle (bakterieller Typ) und hochpotentielle (HiPIP) Ferredoxine unterteilt werden. Ferredoxine mit niedrigem und hohem Potenzial sind durch das folgende Redoxschema miteinander verbunden:
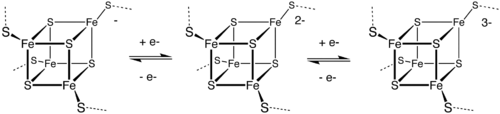
In HiPIP pendelt der Cluster zwischen (Fe4S42+) und (Fe4S43+). Die Potentiale für dieses Redoxpaar reichen von 0,4 bis 0,1 V. In den bakteriellen Ferredoxinen sind die Oxidationszustandspaare (Fe4S4 +) und (Fe4S42 +). Die Potentiale für dieses Redoxpaar reichen von -0,3 bis -0,7 V. Die beiden Familien von 4Fe–4S-Clustern teilen sich die Oxidationsstufe Fe4S42 +. Der Unterschied in den Redoxpaaren ist auf den Grad der Wasserstoffbindung zurückzuführen, der die Basizität der Cysteinylthiolatliganden stark modifiziert. Ein weiteres Redoxpaar, das noch reduzierender als die bakteriellen Ferredoxine ist, ist an der Nitrogenase beteiligt.
Einige 4Fe-4S-Cluster binden Substrate und werden daher als Enzym-Cofaktoren klassifiziert. In der Aconitase bindet der Fe-S-Cluster Aconitat an das eine Fe-Zentrum, dem ein Thiolatligand fehlt. Der Cluster unterliegt keinem Redox, sondern dient als Lewis-Säure-Katalysator zur Umwandlung von Citrat in Isocitrat. In radikalen SAM-Enzymen bindet und reduziert der Cluster S-Adenosylmethionin, um ein Radikal zu erzeugen, das an vielen Biosynthesen beteiligt ist.
3Fe–4S clusterbearbeiten
Es ist auch bekannt, dass Proteine Zentren enthalten, die ein Eisen weniger aufweisen als die üblicheren Kerne. Drei Sulfidionen überbrücken jeweils zwei Eisenionen, während das vierte Sulfid drei Eisenionen überbrückt. Ihre formalen Oxidationsstufen können von + (all-Fe3 + −Form) bis 2- (all-Fe2 + -Form) variieren. In einer Reihe von Eisen–Schwefel-Proteinen kann der Cluster durch Oxidation und Verlust eines Eisenions reversibel in einen Cluster umgewandelt werden. Z.B. besitzt die inaktive Form von Aconitase an und wird durch Zugabe von Fe2 + und Reduktionsmittel aktiviert.
Andere Fe–S-Clusterbearbeiten
Komplexere polymetallische Systeme sind üblich. Beispiele umfassen sowohl die 8Fe- als auch die 7Fe-Cluster in der Nitrogenase. Kohlenmonoxid-Dehydrogenase und die -Hydrogenase weisen ebenfalls ungewöhnliche Fe-S-Cluster auf. Ein spezieller 6-Cystein-koordinierter Cluster wurde in sauerstofftoleranten membrangebundenen Hydrogenasen gefunden.