- Wirtsspezifische Interaktion zwischen dem Rhizobium und Pflanzenpartnern
- Determinierte und unbestimmte Knötchenentwicklung
- Das Wachstum symbiotischer Zellen beinhaltet die Amplifikation des Wirtsgenoms durch Endoreduplikationszyklen
- Verschiedene Schicksale stickstofffixierender Bakteroide
- Wirtspeptide regeln die bakterioide Differenzierung
- NCR247: Ein Beispiel für Multi-Target-Host-Effektor
- Fazit
- Erklärung zum Interessenkonflikt
- Danksagung
Wirtsspezifische Interaktion zwischen dem Rhizobium und Pflanzenpartnern
Die Bakterien, die eine stickstofffixierende Symbiose mit Hülsenfrüchten eingehen, die zu verschiedenen Gruppen von α- und β-Proteobakterien gehören, werden zusammen als Rhizobien bezeichnet (Chen et al., 2003; MacLean et al., 2007). Viele α-Proteobakterien sind an langfristigen Wechselwirkungen mit höheren Eukaryoten beteiligt. Diese Wechselwirkungen reichen von der Oberflächenbesiedlung über fakultative symbiotische Beziehungen bis hin zu obligaten intrazellulären Pathogenen oder Endosymbionten. Die symbiotischen Gene, die für die Knötchenbildung, die Infektion der Wirtszellen und die Stickstofffixierung erforderlich sind, wurden durch lateralen Gentransfer erworben, der die Hauptquelle für die genetische Vielfalt der Rhizobie darstellt. Daher könnte Rhizobie enger mit Krankheitserregern (wie Agrobacterium oder Brucella) verwandt sein als miteinander. Rhizobien neigen dazu, große Genome zu haben (bis zu 10.5 Mbp), die in schnell wachsenden Rhizobien auf multiplen Replikonen dispergiert sind (MacLean et al., 2007). Zum Beispiel hat Sinorhizobium meliloti, der Endosymbiont der Medicago-Spezies, ein dreigeteiltes Genom; ein 3,65 Mbp Chromosom und zwei Megaplasmide, pSymA und pSymB (1,35 und 1,68 Mbp), die beide unentbehrlich sind und die Mehrheit der symbiotischen Gene tragen. Viele S. meliloti-Stämme enthalten jedoch weitere mittelgroße Hilfsplasmide, so dass das S. meliloti-Genom bis zu 9.000 Gene enthalten kann (Barnett et al., 2001; Capela et al., 2001). Im Gegensatz zur Rhizobie besitzen obligate Endosymbionten von Insekten normalerweise ein stark reduziertes (160-450 Kbp) Genom, das ihre Vermehrung sicherstellt und für einige spezifische Biosynthesewege kodiert, einschließlich derjenigen, die den Bedarf des Wirts befriedigen (Moran et al., 2008; Preis et al., 2011). Diese unglaublich reduzierten Genome werden dennoch amplifiziert, um das verminderte Genom mit einem polyploiden DNA-Gehalt zu kompensieren.
Die Pflanzenpartner von Rhizobia gehören zur Familie der Leguminosen/Fabaceae. Die stickstofffixierende Symbiose hat sich in mehreren Linien entwickelt, aber nicht alle Hülsenfrüchte bilden eine Symbiose. Bisher sind 12.000 knotige Hülsenfruchtarten bekannt und jede hat ihre eigenen Rhizobiumpartner. Die Symbiose wird durch Stickstoffmangel der Wirtspflanze ausgelöst, die ihren Rhizobiumpartner aus Milliarden von Bakterien in der Rhizosphäre auswählen muss. Dies wird durch die Sekretion von Flavonoid-Signalmolekülen aus der Wurzel erreicht, die als Chemo-Lockstoffe, vor allem aber als Induktoren der Rhizobium-Nodul-Gene wirken (Oldroyd, 2013). Diese Gene werden für die Produktion bakterieller Signalmoleküle benötigt; die Nod-Faktoren (NFs), die das Knotenentwicklungsprogramm in der Wirtspflanze auslösen (Walker und Downie, 2000). Die NFs sind Lipochitooligosaccharidmoleküle, die wirtsspezifische Substitutionen an den terminalen Zuckerresten und charakteristischen Lipidketten tragen, die von Wirtsrezeptoren vom LysM-Typ erkannt werden und sowohl für die Knötchenentwicklung als auch für bakterielle Infektionen erforderlich sind. Interessanterweise arbeitet die alte Symbiose von Landpflanzen mit arbuskulären Mykorrhizapilzen (AM) mit ähnlichen Lipochitooligosaccharid-Signalmolekülen, den Myc-Faktoren, die von ähnlichen, aber unterschiedlichen LysM-Rezeptoren wie dem NFs wahrgenommen werden (Abdel-Lateif et al., 2012; Oldroyd, 2013). Die Myc-Faktoren und NFs aktivieren einen gemeinsamen Signalweg, aber nach der Beteiligung der gemeinsamen symbiotischen Gene, die in Pflanzen konserviert sind, weichen die Wege ab; einer führt zur Nodulation, der andere zur AM-Symbiose.
Pflanzeninfektion und Knötchenbildung sind komplizierte Prozesse; Nod-Faktoren spielen eine unterschiedliche Rolle bei der Knotenorganogenese und der Wurzelhaarinfektion. Darüber hinaus sind neben Nod-Faktoren verschiedene bakterielle Oberflächenpolysaccharide entscheidend für eine effiziente Infektion (Fraysse et al., 2003). Bei den meisten Leguminosen gelangen die Rhizobien über die Wurzelhaare in den Wirt, wo durch Invagination der Plasmamembran ein Infektionsfaden (IT) gebildet wird, der die sich vermehrenden Bakterien enthält und in Richtung Wurzelrinde wächst. Eine weniger häufige und alte Art der Infektion tritt über Risse auf der Wurzeloberfläche bestimmter Leguminosen auf.
Determinierte und unbestimmte Knötchenentwicklung
Die Knötchenentwicklung erfordert eine mitotische Reaktivierung der kortikalen Zellen, was zur Bildung von Knötchenprimordium führt, das sich dann in stickstofffixierende Wurzelknoten differenziert, die in der zentralen Zone einen mikroaeroben Zustand für die Funktion des sauerstoffempfindlichen Nitrogenase-Enzyms in den Bakteroiden bereitstellen. Abhängig von der vorübergehenden und persistierenden Natur der Wirtszell-Proliferation können die Knötchen entweder vom bestimmten oder unbestimmten Typ sein (Terpolilli et al., 2012; Kondorosi et al., 2013). Bestimmte Knötchen haben kein Meristem und enthalten eine homogene Population symbiotischer Zellen. Bestimmte Knötchen entwickeln sich beispielsweise an Phaseolus vulgaris und Lotus japonicus Wurzeln.
Im Gegenteil, die aktive Zellteilung wird in den unbestimmten Knötchen aufrechterhalten. In der apikalen Region (Zone I) befindet sich ein Knotenmeristem, das durch ständige Erzeugung neuer Zellen ein kontinuierliches Wachstum und eine längliche Knotenform hervorruft. Die Zellen, die das Meristem verlassen, teilen sich nicht mehr und treten in eine Differenzierungsphase ein. Der Infektionsfaden setzt die Bakterien in die sistematischen Zellen frei, die sich allmählich entlang der 12-15 Zellschichten der Infektionszone (Zone II) differenzieren, was zur Entwicklung stickstofffixierender symbiotischer Zellen in der Knotenzone III führt (Abbildung 1; Franssen et al., 1992). Medicago sativa, M. truncatula, Vicia sativa und Pisum sativum sind Beispiele für Pflanzen, die unbestimmte Knötchen bilden.
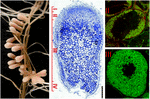
ABBILDUNG 1. Struktur von stickstofffixierenden Wurzelknoten, die in der Symbiose S. meliloti – M. truncatula gebildet wurden. Die verschiedenen Knotenzonen sind auf dem Längsknotenabschnitt angegeben: (I) Meristem, (II) Infektionszone, (III) Stickstofffixierungszone, (IV) Seneszenzzone. Symbiotische Zellen in Zone II enthalten die differenzierenden Endosymbionten, während in Zone III das Wirtszytoplasma vollständig mit langen stickstofffixierenden Bakterioiden gefüllt ist. Mit Syto9 gefärbte Endosymbionten zeigen eine grüne Fluoreszenz.
Das Wachstum symbiotischer Zellen beinhaltet die Amplifikation des Wirtsgenoms durch Endoreduplikationszyklen
Extreme Pflanzenzellvergrößerung kann sowohl in den bestimmten als auch in unbestimmten Knötchen beobachtet werden. Das Zytoplasma einer stickstofffixierenden symbiotischen Zelle beherbergt etwa 50.000 Bakteroide. Um eine so hohe Anzahl von Endosymbionten aufzunehmen, wachsen die Wirtszellen. In M. truncatula-Knötchen ist das Volumen der stickstofffixierenden Zellen 80-fach größer als das der diploiden meristematischen Zellen. Das Wachstum infizierter Zellen erfolgt schrittweise in Zone II und ist die Folge einer wiederholten Endoreduplikation (ER) des Genoms ohne Mitose. In Zone II ist die Zellzyklusmaschinerie noch aktiv, aber der Mangel an mitotischen Cyclinen hemmt die Mitose und wandelt die mitotischen Zyklen in Endoreduplikationszyklen um (Cebolla et al., 1999). Dies wird durch den Zellzyklusschalter CCS52A erreicht, der durch die Zerstörung der mitotischen Cycline wiederholte Runden der Genomduplikation induziert, die zur Bildung allmählich wachsender polyploider Zellen führen (Roudier et al., 2003; Kondorosi und Kondorosi, 2004). Bei Medicago-Arten können die Ploidie-Spiegel 64C erreichen, was einen 64-fach höheren DNA-Gehalt im Vergleich zu den haploiden Zellen darstellt (C entspricht dem haploiden DNA-Gehalt; Vinardell et al., 2003). Die Herunterregulierung von CCS52A in M. truncatula hatte keinen Einfluss auf die Primordiumbildung, war jedoch nachteilig für die Knotendifferenzierung, was darauf hindeutet, dass die ER-Zyklen und die Bildung großer hochpolyploider Zellen für die Knotenfunktion essentiell sind (Vinardell et al., 2003). Interessanterweise sind auch kortikale Zellen, die AM-Pilze enthalten, polyploid, ebenso wie die nematodenspeisenden Riesenwurzelzellen (Favery et al., 2002; In: Genre et al., 2008). In ähnlicher Weise sind auch Insekten-symbiotische Zellen, die Bakteriozyten, die intrazelluläre Endosymbionten beherbergen, groß und polyploid (Nakabachi et al., 2010). In Angiospermenpflanzen ist Polyploidie häufig und das spezifische vererbte Muster der Polyploidie in verschiedenen Organen, Geweben und Zelltypen legt nahe, dass es eine Hauptquelle für die spezialisierte Physiologie von Wirtszellen sein könnte (Nagl, 1976; Edgar et al., 2014). Neben dem Zellwachstum können die multiplen Genkopien und die fehlende Chromosomenkondensation zu höheren Transkriptions- und Stoffwechselaktivitäten beitragen. Die Assoziation von Polyploidie mit verschiedenen Zellfunktionen deutet jedoch auf einen Einfluss von Polyploidie auch auf die Architektur von Nukleosomen und auf das Epigenom hin, das die Aktivierung oder Repression spezifischer Genomregionen steuert. Dementsprechend scheint der polyploide Genomgehalt symbiotischer Zellen eine Voraussetzung für die Knotendifferenzierung und für die Expression der meisten symbiotischen Wirtsgene zu sein (Maunoury et al., 2010).
Verschiedene Schicksale stickstofffixierender Bakteroide
Die aus der IT freigesetzten Bakterien sind im Wirtszytoplasma als organellenartige Strukturen, sogenannte Symbiosomen, vorhanden. Die Bakterien haben keinen direkten Kontakt mit dem Zytoplasma, da sie von einer peribakteroiden Membran umgeben sind, die auch als Symbiosomenmembran (SM) bekannt ist. Das Bakteroid, das SM und der Raum zwischen ihnen bilden das Symbiosom (Catalano et al., 2004). Der SM während seiner Entstehung spiegelt seinen Plasmamembranursprung wider, spätere Modifikationen seiner Zusammensetzung eröffnen neue, spezialisierte Rollen an der Wirt-Endosymbionten-Grenzfläche (Limpens et al., 2009; Ivanov et al., 2012; Brear et al., 2013; Sinharoy et al., 2013). Die Bakteroide vermehren sich in den wachsenden Wirtsknotenzellen bis zu einer bestimmten Zelldichte, passen sich dem endosymbiotischen Lebensstil und den mikroaeroben Bedingungen an und reifen zu stickstofffixierenden Bakteroiden. Die Form und Physiologie von Bakteroiden kann jedoch bei den verschiedenen Leguminosen auffallend unterschiedlich sein. In bestimmten Hülsenfruchtwirten haben die stickstofffixierenden Bakteroide die gleiche Morphologie wie kultivierte Zellen; Diese Art von Bakteroiden kann in die freilebende Form zurückkehren. In anderen Assoziationen werden die Bakteroide irreversibel in polyploide, vergrößerte, nicht kultivierbare Endosymbionten umgewandelt. Diese endständig differenzierten Bakteroide können länglich und sogar verzweigt sein und 5- bis 10-fach länger als die frei lebenden Zellen sein oder je nach Wirt ein 8- bis mindestens 20-fach amplifiziertes Genom aufweisen (Mergaert et al., 2006; Nakabachi et al., 2010). Die terminale Differenzierung von Bakteroiden wird vom Wirt kontrolliert, entwickelt in mehreren Zweigen der Leguminosen-Familie, was auf einen Wirtsvorteil und wahrscheinlich eine höhere symbiotische Leistung hinweist (Oono et al., 2010). Die terminale bakterioide Differenzierung ist am besten in der S. meliloti – M. truncatula Symbiose aufgeklärt. In M. Truncatula-Knötchen, die sichtbarsten Ereignisse der terminalen Bakterioiddifferenzierung, treten in Zone II auf. Die Vermehrung von Bakteroiden stoppt in der Mitte von Zone II, wo die Zellverlängerung und die gleichmäßige Amplifikation der multiplen Replikone durch Endoreduplikationszyklen beginnen. Entlang 2-3 Zellschichten an der Grenze von Zone II und III (genannt Interzone) ist ein plötzliches Wachstum von Bakteroiden sichtbar, das praktisch ihre endgültige Größe erreicht, jedoch findet die Stickstofffixierung nur in Zone III statt.
Wirtspeptide regeln die bakterioide Differenzierung
Der Vergleich von Knotentranskriptomen von Leguminosen mit reversibler und irreversibler bakterioider Differenzierung ergab die Existenz mehrerer Hundert kleiner Gene, die nur im Genom jener Wirtspflanzen vorhanden waren, bei denen die bakterioide Differenzierung terminal war. In M. truncatula produzieren die Knotenzellen mindestens 600 knotenspezifische symbiotische Peptide (symPEPs). Die symPEP-Gene werden nur in den mit S. meliloti infizierten polyploiden symbiotischen Zellen aktiviert (Kevei et al., 2002; Mergaert et al., 2003), jedoch bestimmte Sätze in den früheren, andere in den späteren Stadien der Knötchenentwicklung. Ein großer Teil, mehr als 500 Gene, kodieren für knotenspezifische cysteinreiche (NCR) Peptide (Mergaert et al., 2003; Alunni et al., 2007; Nallu et al., 2014). Die NCR-Peptide zielen auf die Bakteroide ab, und wenn ihre Abgabe an die Endosymbionten blockiert wurde, wurde die Bakterioiddifferenzierung aufgehoben, was zeigt, dass die Peptide für die terminale Differenzierung von S. meliloti-Bakteroiden verantwortlich sind (Van de Velde et al., 2010). Die hohe Sequenzvielfalt und die charakteristischen Expressionsmuster von NCR-Genen deuten auf eine Vielfalt ihrer Funktionen, Wirkweisen und bakteriellen Ziele in verschiedenen Stadien der bakteriellen Reifung hin (Abbildung 2). Warum produziert die Wirtszelle jedoch ein Arsenal an NCRs? Was kann der Vorteil eines so vielfältigen Peptidrepertoires sein? Ist es notwendig für die Interaktion des Wirts mit verschiedenen Bakterien? Die symbiotischen Partner von M. truncatula sind S. meliloti und S. medicae, im Boden gibt es jedoch unzählige Stammvarianten beider Arten. M. truncatula wird auch durch viele verschiedene Ökotypen und Akzessionen repräsentiert, die sich in Anzahl, Sequenzen und Expressionsprofil von NCR-Genen und in ihren symbiotischen Wechselwirkungen mit verschiedenen S. meliloti- und S. medicae-Stämmen unterscheiden (Nallu et al., 2014; Roux et al., 2014). Während ein Knoten einen einzelnen Bakterientyp enthält, können die verschiedenen Knoten auf demselben Wurzelsystem unterschiedliche Bakterienpopulationen besitzen. Es ist möglich, dass die Pflanze, die die verschiedenen Endosymbionten erkennt, sie mit einem stammspezifischen Repertoire an Peptiden manipuliert. Diese Unterschiede können eine zusätzliche Kontrollebene für die Wirt-Symbionten-Spezifität und damit für die Knoteneffizienz hinzufügen.

ABBILDUNG 2. Differentielle Expression von symPEP-Genen in M. truncatula-Knötchen. Schwarzes Signal: In-situ-Hybridisierung, blaues Signal: GUS-Aktivität von symPEP-Promotor-GUS-Fusionen in transgenen Knötchen.
Obwohl AMPS einzigartige Peptidklassen darstellen, ähneln ihre Strukturen antimikrobiellen Peptiden (AMPs). AMPs mit einem breiten Spektrum an mikrobieller zelltötender Aktivität sind am häufigsten kationisch und provozieren den Zelltod durch Porenbildung, Membranzerstörung und konsequente Lyse von mikrobiellen Zellen. Die Tatsache, dass die Zellteilungsfähigkeit während der Endosymbiontendifferenzierung definitiv verloren geht, deutet darauf hin, dass zumindest bestimmte symPEPs antimikrobielle Aktivitäten aufweisen. Die Behandlung von Bakterien mit synthetischen kationischen NCRs führte in der Tat zu einer schnellen und effizienten dosisabhängigen Elimination verschiedener gramnegativer und grampositiver Bakterien, einschließlich wichtiger menschlicher und pflanzlicher Krankheitserreger (Van de Velde et al., 2010; Tiricz et al., 2013). Dieser Ex-Planta-Abtötungseffekt korrelierte jedoch mit der Permeabilisierung mikrobieller Membranen, da Bakterien in ihrer natürlichen Umgebung – in den Knötchenzellen – die Bakterienmembranen nicht permeabilisieren und die Endosymbionten nicht abtöten. Höchstwahrscheinlich sind die Peptidkonzentrationen in den Knötchen signifikant niedriger als die in den In-vitro-Assays angewandten. Darüber hinaus werden kationische Peptide zusammen mit anionischen und neutralen Peptiden in derselben Zelle hergestellt, und eine mögliche Kombination von einigen zehn oder Hunderten von Peptiden mit unterschiedlicher Ladung und Hydrophobie könnte die direkte bakterizide Wirkung der kationischen Peptide neutralisieren.
Die Beteiligung von AMPs oder AMP-ähnlichen Peptiden ist für die Rhizobium-Leguminosen-Symbiose nicht einzigartig. Im Rüsselkäfer Sitophilus produzieren die symbiotischen Zellen das antimikrobielle Peptid Coleoptericin-A (ColA), das durch Hemmung der Zellteilung die Entwicklung riesiger filamentöser Endosymbionten provoziert und das benachbarte Insektengewebe vor bakterieller Invasion schützt (Login et al., 2011). In diesem System ist ein einzelnes Peptid ausreichend für die Differenzierung des obligaten vertikal übertragenen Endosymbionten im Gegensatz zu Knötchen, die mit Hunderten von symPEPs arbeiten und unzählige Stammvarianten als ihre Endosymbionten beherbergen können. In der Blattlaus-Buchnera-Symbiose produzieren die Wirtszellen auch bakteriozytenspezifische Peptide, einschließlich cysteinreicher Peptide (BCR), die den Medicago-NCR-Peptiden ähneln, jedoch wurden die Funktionen dieser symbiotischen Peptide noch nicht berichtet (Shigenobu und Stern, 2013).
NCR247: Ein Beispiel für Multi-Target-Host-Effektor
Transkriptom-Analyse von M. Truncatula-Knötchen in verschiedenen Stadien ihrer Entwicklung, Laser-Mikrodissektion von Knötchenregionen, In-situ-Hybridisierung, Immunlokalisation ausgewählter Peptide und symPEP-Promotor-Reporter-Genfusionen in transgenen Knötchen ermöglichen die Kartierung der Wirkung einzelner Peptide in den symbiotischen Zellen von der frühen Infektion bis zum späten Stickstofffixierungszustand. NCR247 wird in den älteren Zellschichten der Zone II und in der Interzone exprimiert, wo die bakterielle Zellteilung aufhört und eine bemerkenswerte Dehnung der Endosymbionten auftritt (Farkas et al., 2014). Dieses kleine kationische Peptid tötete effektiv verschiedene Mikroben in vitro und die In-Silico-Analyse zeigte seine extremen Proteinbindungskapazitäten. FITC-markiertes NCR247 gelangte in das bakterielle Cytosol, wo seine Wechselwirkungen mit zahlreichen bakteriellen Proteinen möglich waren. Bindungspartner wurden durch Behandlung von S. meliloti-Bakterien oder Bakteroiden mit StrepII / FLAG-markierten Peptiden identifiziert, gefolgt von Affinitätschromatographie und Identifizierung von Interaktionspartnern mit LC-MS / MS und Western-Analyse (Farkas et al., 2014).
Einer der Interaktoren war das FtsZ-Zellteilungsprotein, das eine entscheidende primäre Rolle bei der Zellteilung spielt. Es ist bekannt, dass eine Reihe von antibiotischen Peptiden durch die Wechselwirkung mit FtsZ eine bakterizide oder bakteriostatische Wirkung ausübt, deren Polymerisation hemmt und dadurch die richtige Z-Ring- und Septumbildung behindert (Handler et al., 2008). NCR247 wurde zusammen mit FtsZ aus dem bakteriellen Zytoplasma gereinigt und es wurde gezeigt, dass es die Septumbildung stört. NCR035, das in vitro auch bakterizide Wirkung zeigt und in den gleichen symbiotischen Zellen produziert wird, wie NCR247 am Teilungsseptum akkumuliert, was auf eine gleichzeitige oder aufeinanderfolgende Wirkung dieser Peptide und die Entwicklung mehrerer Wirtsstrategien zur Hemmung der Endosymbiontenproliferation hinweist. Eine weitere Studie zeigte, dass die Expression wichtiger Zellteilungsgene, einschließlich der für die Z-Ring-Funktion erforderlichen Gene, in Zellen, die mit NCR247 behandelt wurden, stark abgeschwächt war (Penterman et al., 2014). Die Vorbehandlung von Bakterien mit sub-letalen NCR247-Konzentrationen schaffte die Lokalisation von FITC-NCR035 im Septum ab und provozierte eine Zellverlängerung (Farkas et al., 2014).
Ribosomale Proteine waren die am häufigsten vorkommenden NCR247-Interaktionspartner. Es wurde beobachtet, dass NCR247 die bakterielle Proteinsynthese sowohl in vivo als auch in vitro dosisabhängig stark hemmt (Farkas et al., 2014). Diese Ergebnisse deuteten darauf hin, dass ein Modus der NCR247-Peptidwirkung sowohl in Bakterienzellen als auch in Bakterioiden an die Ribosomen bindet. Interessanterweise wurden ein verändertes Muster und eine reduzierte Komplexität der interagierenden Proteine in den Bakteroiden beobachtet. Dementsprechend war das allgemeine Expressionsniveau von ribosomalen Proteinen in den Bakteroiden im Durchschnitt 20-fach niedriger als in den frei lebenden Zellen mit unterschiedlicher relativer Häufigkeit von Transkripten einzelner ribosomaler Proteine. Die Diversifizierung der Ribosomen in Bakterioiden kann eine bedeutende Rolle spielen, indem sie zur fortgeschrittenen Translation spezifischer Proteine beiträgt und dadurch die spezialisierte, energieintensive Physiologie der hochgradig vorhandenen Stickstofffixierungsfunktion unterstützt.
Das GroEL-Chaperon war auch ein direkter Interaktionspartner von NCR247 (Farkas et al., 2014). Von den 5 GroEL-Proteinen reicht GroEL1 oder GroEL2 zum Überleben aus, während GroEL1, das in hohem Maße im Knoten exprimiert wird, für die Symbiose essentiell ist (Bittner et al., 2007). Es wird für die vollständige Aktivierung der Knotengene und den Aufbau des Nitrogenase-Komplexes benötigt. GroEL besitzt eine extreme funktionelle Vielseitigkeit durch Interaktion mit Hunderten von Proteinen. Die NCR247-GroEL1-Wechselwirkung kann Konsequenzen direkt auf GroEL, aber indirekt auch auf die GroEL-Substrate und die damit verbundenen biologischen Prozesse haben. Das Fehlen von GroEL1 beeinträchtigte die bakterielle Infektion und die Aufrechterhaltung und Differenzierung von Bakteroiden, was einen allgemeinen Bedarf an GroEL1 in allen Stadien der stickstofffixierenden Knötchenentwicklung zeigt.
Die Beteiligung von GroEL- und Wirtspeptiden an Mikroben-Wirt-Interaktionen ist für die Rhizobium-Leguminosen-Symbiose nicht einzigartig. In den symbiotischen Zellen des Rüsselkäfers interagiert Coleoptericin-A (ColA) auch mit GroEL (Login et al., 2011). GroEL spielt auch eine wichtige Rolle bei der Aufrechterhaltung von Endosymbionten (Moran, 1996; Kupper et al., 2014). Da die meisten symbiotischen Systeme noch unerforscht sind und genomische und proteomische Werkzeuge mit hohem Durchsatz erst seit kurzem verfügbar sind, Wir können nur vorhersagen, dass die Differenzierung von Wirtspeptiden durch Endosymbionten, ebenso die Genomamplifikation von Wirtszellen und terminal differenzierte Endosymbionten sind allgemeine Strategien der Symbiose.
Fazit
Symbiotische und pathogene Bakterien verwenden ähnliche Ansätze, um mit ihren Wirten zu interagieren und in Wirtszellen zu überleben, auch wenn die Ergebnisse dieser Interaktionen auffallend unterschiedlich sind. Pflanzen und Tiere können bei der Wahrnehmung von mAmps (Microorganism-associated molecular Patterns) angeborene Immunantworten auf Mikroorganismen erzeugen. Diese Wahrnehmung führt zur Aktivierung von Signalkaskaden und zur Produktion antimikrobieller Effektoren. AMP-ähnliche Wirtspeptide wie das M. Truncatula-NCR-Peptide oder das antimikrobielle Peptid Weevil ColA spielen eine zentrale und facettenreiche Rolle bei der Kontrolle der Vermehrung und Differenzierung von Endosymbionten, wodurch das Vorhandensein von Bakterien auf die symbiotischen Zellen beschränkt wird. Daher nutzen Wirtsorganismen diese Effektorpeptide, um ausgewählte mikrobielle Eindringlinge zu zähmen und sogar für den Dienst einzustellen.
Erklärung zum Interessenkonflikt
Die Autoren erklären, dass die Forschung in Abwesenheit von kommerziellen oder finanziellen Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Danksagung
Die Arbeit in unseren Laboren wird durch den „SYM-BIOTICS“ Advanced Grant des Europäischen Forschungsrates an Éva Kondorosi (Grant-Nummer 269067) und durch TÁMOP-4.2.2 unterstützt.A-11/1/KONV-2012-0035 gefördert durch die Europäische Union und kofinanziert durch den Europäischen Sozialfonds.
Abdel-Lateif, K., Bogusz, D. und Hocher, V. (2012). Die Rolle von Flavonoiden bei der Etablierung von Pflanzenwurzelendosymbiosen mit arbuskulären Mykorrhiza-Pilzen, Rhizobien und Frankia-Bakterien. Pflanzen Signal. Verhalten. 7, 636–641. Ursprungsbezeichnung: 10.4161/psb.20039
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Alunni, B., Kevei, Z., Redondo-Nieto, M., Kondorosi, A., Mergaert, P., and Kondorosi, E. (2007). Genomic organization and evolutionary insights on GRP and NCR genes, two large nodule-specific gene families in Medicago truncatula. Mol. Plant Microbe Interact. 20, 1138–1148. doi: 10.1094/MPMI-20-9-1138
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Barnett, M. J., Fisher, R. F., Jones, T., Komp, C., Abola, A. P., Barloy-Hubler, F.,et al. (2001). Nukleotidsequenz und vorhergesagte Funktionen des gesamten Sinorhizobium meliloti pSymA Megaplasmid. Prok. Natl. Acad. Sci. USA 98, 9883-9888. doi: 10.1073/pnas.161294798
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Bittner, A. N., Foltz, A. und Oke, V. (2007). Nur eines von fünf GroEL-Genen ist für die Lebensfähigkeit und erfolgreiche Symbiose in Sinorhizobium meliloti erforderlich. In: J. Bacteriol. 189, 1884–1889. Ursprungsbezeichnung: 10.1128/JB.01542-06
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Brear, E. M., Day, D. A., and Smith, P. M. (2013). Iron: an essential micronutrient for the legume-Rhizobium symbiosis. Front. Plant Sci. 4:359. doi: 10.3389/fpls.2013.00359
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Catalano, C. M., Lane, W. S., and Sherrier, D. J. (2004). Biochemical characterization of symbiosome membrane proteins from Medicago truncatula root nodules. Electrophoresis 25, 519–531. doi: 10.1002/elps.200305711
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Capela, D., Barloy-Hubler, F., Gouzy, J., Bothe, G., Ampe, F., Batut, J.,et al. (2001). Analyse der Chromosomensequenz des Hülsenfruchtsymbionten Sinorhizobium meliloti Stamm 1021. Prok. Natl. Acad. Sci. USA 98, 9877-9882. doi: 10.1073/pnas.161294398
Pubmed Zusammenfassung | Pubmed Volltext / CrossRef Volltext
Cebolla, A., Vinardell, J. M., Kiss, E., Oláh, B., Roudier, F., Kondorosi, A.,et al. (1999). Der mitotische Inhibitor ccs52 wird zur Endoreduplikation und ploidieabhängigen Zellvergrößerung in Pflanzen benötigt. EMBO J. 18, 4476-4484. doi: 10.1093/emboj/18.16.4476
CrossRef Volltext
Chen, W. M., Moulin, L., Bontemps, C., Vandamme, P., Béna, G. und Boivin-Masson, C. (2003). Die symbiotische Stickstofffixierung von Hülsenfrüchten durch β-Proteobakterien ist in der Natur weit verbreitet. In: J. Bacteriol. 185, 7266–7272. Ursprungsbezeichnung: 10.1128/JB.185.24.7266-7272.2003
CrossRef Volltext
Edgar, B. A., Zielke, N. und Gutierrez, C. (2014). Endocycles: eine wiederkehrende evolutionäre Innovation für das postmitotische Zellwachstum. Nat. In: Rev. Mol. Zelle. Biol. 15, 197–210. doi: 10.1038/nrm3756
Pubmed Zusammenfassung | Pubmed Volltext / CrossRef Volltext
Farkas, A., Maróti, G., Dürgõ, H., Györgypál, Z., Lima, R. M., Medzihradszky, K. F.,et al. (2014). Das symbiotische Peptid NCR247 von Medicago truncatula trägt durch mehrere Mechanismen zur Differenzierung von Bakterien bei. Prok. Natl. Acad. Sci. U.S.A. 111, 5183-5188. doi: 10.1073/pnas.1404169111
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Favery, B., Complainville, A., Vinardell, J. M., Lecomte, P., Vaubert, D., Mergaert, P.,et al. (2002). The endosymbiosis-induced genes ENOD40 and CCS52a are involved in endoparasitic-nematode interactions in Medicago truncatula. Mol. Plant-Microbe Interact. 15, 1008–1013. doi: 10.1094/MPMI.2002.15.10.1008
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Franssen, H. J., Vijn, I., Yang, W. C., and Bisseling, T. (1992). Developmental aspects of the Rhizobium-legume symbiosis. Plant Mol. Biol. 19, 89–107. doi: 10.1007
CrossRef Full Text
Fraysse, N., Couderc, F., and Poinsot, V. (2003). Surface polysaccharide involvement in establishing the Rhizobium–legume symbiosis. Eur. J. Biochem. 270, 1365–1380. doi: 10.1046/j.1432-1033.2003.03492.x
CrossRef Full Text
Genre, A., Chabaud, M., Faccio, A., Barker, D. G., and Bonfante, P. (2008). Die Anordnung der Präpenetrationsapparate geht den Kolonisationsmustern von arbuskulären Mykorrhizapilzen im Wurzelkortex von Medicago truncatula und Daucus carota voraus und sagt diese voraus. Pflanzenzelle 20, 1407-1420. ursprungsbezeichnung: 10.1105/tpc.108.059014
Pubmed Zusammenfassung | Pubmed Volltext / CrossRef Volltext
Handler, A. A., Lim, J. E. und Losick, R. (2008). Peptid-Inhibitor der Zytokinese während der Sporulation in Bacillus subtilis. Mol. Microbiol. 68, 588–599. Ursprungsbezeichnung: 10.1111/j.1365-2958.2008.06173.x
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Ivanov, S., Fedorova, E. E., Limpens, E., De Mita, S., Genre, A., Bonfante, P.,et al. (2012). Rhizobium–legume symbiosis shares an exocytotic pathway required for arbuscule formation. Proc. Natl. Acad. Sci. U.S.A. 109, 8316–8321. doi: 10.1073/pnas.1200407109
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Kevei, Z., Vinardell, J. M., Kiss, G. B., Kondorosi, A., and Kondorosi, E. (2002). Glycinreiche Proteine, die von einer knotenspezifischen Genfamilie kodiert werden, sind an verschiedenen Stadien der symbiotischen Knotenentwicklung in Medicago spp. beteiligt. Mol. Pflanzenmikroben interagieren. 15, 922–931. doi: 10.1094/MPMI.2002.15.9.922
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Kondorosi, E. und Kondorosi, A. (2004). Endoreduplikation und Aktivierung des Anaphase-fördernden Komplexes während der symbiotischen Zellentwicklung. FEBS Lett. 567, 152–157. doi: 10.1016/j.febslet.2004.04.075
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Kondorosi, E., Mergaert, P. und Kereszt, A. (2013). Ein Paradigma für endosymbiotisches Leben: Zelldifferenzierung von Rhizobiumbakterien, hervorgerufen durch Wirtspflanzenfaktoren. Jahr. Rev. Microbiol. 67, 611–628. doi: 10.1146/annurev-micro-092412-155630
CrossRef Volltext
Kupper, M., Gupta, S. K., Feldhaar, H. und Gross, R. (2014). Vielseitige Rollen des Chaperonin GroEL in Mikroorganismen-Insekten-Interaktionen. FEMS Microbiol. Lett. 353, 1–10. doi: 10.1111/1574-6968.12390
Pubmed Abstract | Pubmed Full Text / CrossRef Full Text
Limpens, E., Ivanov, S., van Esse, W., Voets, G., Fedorova, E. und Bisseling, T. (2009). Medicago N2-fixierende Symbiosomen erwerben den endozytären Identitätsmarker Rab7, verzögern jedoch den Erwerb der vakuolären Identität. Pflanzenzelle 21, 2811-2828. ursprungsbezeichnung: 10.1105/tpc.108.064410
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Login, F. H., Balmand, S., Vallier, A., Vincent-Monégat, C., Vigneron, A., Weiss-Gayet, M.,et al. (2011). Antimikrobielle Peptide halten Insektenendosymbionten unter Kontrolle. Wissenschaft 334, 362-365. doi: 10.1126/Wissenschaft.1209728
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
MacLean, A. M., Finan, T. M. und Sadowsky, M. J. (2007). Genome der symbiotischen stickstofffixierenden Bakterien von Leguminosen. In: Plant Physiol. 144, 615–622. doi: 10.1104/pp.107.101634
Pubmed Zusammenfassung | Pubmed Volltext | CrossRef Volltext
Maunoury, N., Redondo-Nieto, M., Bourcy, M., Van de Velde, W., Alunni, B., Laporte, P.,et al. (2010). Differenzierung von symbiotischen Zellen und Endosymbionten werden an zwei Transkriptom-Schalter in Medicago truncatula Nodulation gekoppelt. Plus EINS 5:e9519. doi: 10.1371/Zeitschrift.pone.0009519
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Mergaert, P., Nikovics, K., Kelemen, Z., Maunoury, N., Vaubert, D., Kondorosi, A.,et al. (2003). Eine neuartige Familie in Medicago truncatula, bestehend aus mehr als 300 knötchenspezifischen Genen, die für kleine, sezernierte Polypeptide mit konservierten Cysteinmotiven kodieren. In: Plant Physiol. 132, 161–173. doi: 10.1104/pp.102.018192
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Mergaert, P., Uchiumi, T., Alunni, B., Evanno, G., Cheron, A., Catrice, O.,et al. (2006). Eukaryotic control on bacterial cell cycle and differentiation in the Rhizobium-legume symbiosis. Proc. Natl. Acad. Sci. U.S.A. 103, 5230–5235. doi: 10.1073/pnas.0600912103
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Moran, N. A. (1996). Beschleunigte Evolution und Muller-Ratsche in endosymbiotischen Bakterien. Prok. Natl. Acad. Sci. USA 93, 2873-2878. doi: 10.1073/pnas.93.7.2873
CrossRef Volltext
Moran, N. A., McCutcheon, J. P. und Nakabachi, A. (2008). Genomik und Evolution vererbbarer bakterieller Symbionten. Jahr. In: Rev. Genet. 42, 165–690. doi: 10.1146/Jahrbuch.Genet.41.110306.130119
CrossRef Volltext
Nakabachi, A., Koshikawa, S., Miura, T. und Miyagishima, S. (2010). Genomgröße von Pachypsylla venusta (Hemiptera: Psyllidae) und die Ploidie seiner Bakteriozyten, der symbiotischen Wirtszelle, die intrazelluläre mutualistische Bakterien mit dem kleinsten zellulären Genom beherbergt. Bull. Entomol. 100, 27-33. doi: 10.1017/S0007485309006737
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Nagl, W. (1976). DNA-Endoreduplikation und Polytenie als evolutionäre Strategien verstanden. Natur 261, 614-615. doi: 10.1038/261614a0
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Nallu, S., Silverstein, K. A., Zhou, P., Young, ND und Vandenbosch, KA (2014). Divergenzmuster einer großen Familie von cysteinreichen Knötchenpeptiden in Akzessionen von Medicago truncatula. Pflanze J. 78, 697-705. doi: 10.1111/tpj.12506
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Oldroyd, G. E. (2013). Speak, friend, and enter: Signalsysteme, die nützliche symbiotische Assoziationen in Pflanzen fördern. Nat. Rev. Microbiol. 11, 252–263. doi: 10.1038/nrmicro2990
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Oono, R., Schmitt, I., Sprent, J. I., and Denison, R. F. (2010). Multiple evolutionary origins of legume traits leading to extreme rhizobial differentiation. New Phytol. 187, 508–520. doi: 10.1111/j.1469-8137.2010.03261.x
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Penterman, J., Abo, R. P., De Nisco, N. J., Arnold, M. F., Longhi, R., Zanda, M.,et al. (2014). Wirtspflanzenpeptide lösen eine transkriptionelle Reaktion aus, um den Sinorhizobium meliloti-Zellzyklus während der Symbiose zu steuern. Prok. Natl. Acad. Sci. USA 111, 3561-3566. doi: 10.1073/pnas.1400450111
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Price, D. R., Duncan, R. P., Shigenobu, S. und Wilson, A. C. (2011). Genomexpansion und differentielle Expression von Aminosäuretransportern an der symbiotischen Grenzfläche Blattlaus / Buchnera. Mol. Biol. In: Evol. 28, 3113–3126. doi: 10.1093/molbev/msr140
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Roudier, F., Fedorova, E., Lebris, M., Lecomte, P., Györgyey, J., Abad, P.,et al. (2003). The Medicago species A2-type cyclin is auxin regulated and involved in meristem formation but dispensable for endoreduplication-associated developmental programs. Plant Physiol. 131, 1091–1103. doi: 10.1104/pp.102.011122
CrossRef Full Text
Roux, B., Rodde, N., Jardinaud, M. F., Timmers, T., Sauviac, L., Cottret, L.,et al. (2014). Eine integrierte Analyse der pflanzlichen und bakteriellen Genexpression in symbiotischen Wurzelknoten mittels Laser-Capture-Mikrodissektion in Verbindung mit RNA-Sequenzierung. Pflanze J. 77, 817-837. doi: 10.1111/tpj.12442
Pubmed Zusammenfassung | Pubmed Volltext / CrossRef Volltext
Shigenobu, S. und Stern, D. L. (2013). Blattläuse entwickelten neuartige sekretierte Proteine für die Symbiose mit bakteriellen Endosymbionten. Prok. Biol. Sci. 280, 20121952. Ursprungsbezeichnung: 10.1098/rspb.2012.1952
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Sinharoy, S., Torres-Jerez, I., Bandyopadhyay, K., Kereszt, A., Pislariu, C. I., Nakashima, J.,et al. (2013). Der C2H2-Transkriptionsfaktor-Regulator der Symbiosomen-Differenzierung unterdrückt die Transkription des sekretorischen Signalweg-Gens VAMP721a und fördert die Symbiosomen-Entwicklung in Medicago truncatula. Pflanzenzelle 25, 3584-3601. ursprungsbezeichnung: 10.1105/tpc.113.114017
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Terpolilli, J. J., Hood, G. A., and Poole, P. S. (2012). What determines the efficiency of N(2)-fixing Rhizobium-legume symbioses? Adv. Microb. Physiol. 60, 325–389. doi: 10.1016/B978-0-12-398264-3.00005-X
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Tiricz, H., Szûcs, A., Farkas, A., Pap, B., Lima, R. M., Maróti, G.,et al. (2013). Antimicrobial nodule-specific cysteine-rich peptides induce membrane depolarization associated changes in Sinorhizobium meliloti. Appl. Env. Microbiol. 79, 6737–6746. doi: 10.1128/AEM.01791-13
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Van de Velde, W., Zehirov, G., Szatmari, A., Debreczeny, M., Ishihara, H., Kevei, Z.,et al. (2010). Plant peptides govern terminal differentiation of bacteria in symbiosis. Science 327, 1122–1126. doi: 10.1126/science.1184057
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Vinardell, J. M., Fedorova, E., Cebolla, A., Kevei, Z., Horvath, G., Kelemen, Z.,et al. (2003). Die Endoreduplikation, die durch den Anaphase-fördernden komplexen Aktivator CCS52A vermittelt wird, ist für die symbiotische Zelldifferenzierung in Medicago truncatula-Knötchen erforderlich. Pflanzenzelle 15, 2093-2105. ursprungsbezeichnung: 10.1105/tpc.014373
Pubmed Zusammenfassung | Pubmed Volltext / CrossRef Volltext
Walker, S. A., und Downie, J. A. (2000). Eintrag von Rhizobium leguminosarum bv. viciae in Wurzelhaare erfordert minimale Nickenfaktor Spezifität, aber nachfolgende Infektion Fadenwachstum erfordert nodO oder Knoten. Mol. Pflanze-Mikrobe interagieren.13, 54–62. doi: 10.1094/MPMI.2000.13.7.754
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text