- Interacción Específica del huésped Entre el Rizobio y los Socios de la Planta
- Desarrollo de nódulos determinado e Indeterminado
- El crecimiento de Células Simbióticas Implica la Amplificación del Genoma del Huésped mediante Ciclos de Endoreduplicación
- Diferentes Destinos de Bacteroides Fijadores de Nitrógeno
- Los péptidos Huésped Gobiernan la diferenciación bacteroide
- NCR247: Un ejemplo para el análisis del transcriptoma del Efector de Host Multiobjetivo
- Conclusión
- Declaración de Conflicto de Intereses
- Reconocimientos
Interacción Específica del huésped Entre el Rizobio y los Socios de la Planta
Las bacterias que forman simbiosis fijadora de nitrógeno con plantas de leguminosas pertenecientes a diversos grupos de proteobacterias α y β se denominan colectivamente rizobias (Chen et al., 2003; MacLean et al., 2007). Muchas proteobacterias α participan en interacciones a largo plazo con eucariotas superiores. Estas interacciones van desde la colonización superficial a través de relaciones simbióticas facultativas hasta estilos de vida endosimbiontes o patógenos intracelulares obligatorios. Los genes simbióticos necesarios para la formación de nódulos, la infección de células huésped y la fijación de nitrógeno se han adquirido mediante transferencia lateral de genes, que es la fuente principal de diversidad genética de los rizobios. Por lo tanto, la rizobia podría estar más estrechamente relacionada con patógenos (como Agrobacterium o Brucella) que entre sí. Los rizobios tienden a tener genomas grandes (hasta 10.5 Mbp) que en rizobias de rápido crecimiento se dispersan en múltiples replicones (MacLean et al., 2007). Por ejemplo, Sinorhizobium meliloti, el endosimbionte de la especie Medicago, tiene un genoma dividido en tres partes; un cromosoma de 3,65 Mbp y dos megaplásmidos, pSymA y pSymB (1,35 y 1,68 Mbp), ambos indispensables y portadores de la mayoría de genes simbióticos. Sin embargo, muchas cepas de S. meliloti contienen plásmidos auxiliares de tamaño medio y, por lo tanto, el genoma de S. meliloti puede contener hasta 9.000 genes (Barnett et al., 2001; Capela et al., 2001). En contraste con la rizobia, los endosimbiontes obligados de los insectos generalmente poseen un genoma fuertemente reducido (160-450 Kbp) que asegura su multiplicación y codifica para unas pocas vías biosintéticas específicas, incluidas aquellas que satisfacen las necesidades del huésped (Moran et al., 2008; Price et al., 2011). Estos genomas increíblemente reducidos, sin embargo, se amplifican compensando el genoma disminuido con un contenido de ADN poliploide.
Las plantas asociadas de rhizobia pertenecen a la familia Leguminosae/Fabaceae. La simbiosis fijadora de nitrógeno ha evolucionado en varios linajes, pero no todas las legumbres forman simbiosis. Hasta ahora se conocen 12.000 especies de leguminosas noduladas y cada una tiene su(s) compañero (s) de Rizobio. La simbiosis se desencadena por la inanición de nitrógeno de la planta huésped que tiene que seleccionar a su compañero de Rizobio de miles de millones de bacterias en la rizosfera. Esto se logra mediante la secreción de moléculas de señal de flavonoides de la raíz que actúan como quimiotractantes pero, lo que es más importante, como inductores de los genes de nodulación de Rizobio (Oldroyd, 2013). Estos genes son necesarios para la producción de moléculas de señal bacteriana; los factores de Nod (NFs) que desencadenan el programa de desarrollo de nódulos en la planta huésped (Walker y Downie, 2000). Los NFs son moléculas de lipochitooligosacáridos que transportan sustituciones específicas del huésped en los residuos terminales de azúcar y las cadenas lipídicas características, que son reconocidas por los receptores del huésped tipo LysM y son necesarias tanto para el desarrollo de nódulos como para la infección bacteriana. Curiosamente, la antigua simbiosis de plantas terrestres con hongos micorrícicos arbusculares (AM) opera con moléculas de señal de lipochitooligosacáridos similares, los factores Myc que son percibidos por receptores de tipo LysM similares pero diferentes como los NFs (Abdel-Lateif et al., 2012; Oldroyd, 2013). Los factores Myc y NFs activan una vía de señalización común, pero después de la participación de los genes simbióticos comunes conservados en las plantas, las vías se desvían; una conduce a la nodulación, la otra a la simbiosis AM.
La infección de plantas y la formación de nódulos son procesos intrincados; Los factores de inclinación de cabeza desempeñan papeles distintos en la organogénesis de los nódulos y en la infección del vello radicular. Además, además de los factores de Nod, varios polisacáridos de superficie bacteriana son cruciales para una infección eficiente(Fraysse et al., 2003). En la mayoría de las legumbres, los rizobios entran en el huésped a través de los pelos de la raíz, donde por invaginación de la membrana plasmática se forma un hilo de infección (IT) que contiene las bacterias multiplicadoras y crece hacia la corteza de la raíz. Un modo de infección menos frecuente y antiguo ocurre a través de grietas en la superficie de la raíz de ciertas legumbres.
Desarrollo de nódulos determinado e Indeterminado
El desarrollo de nódulos requiere la reactivación mitótica de las células corticales que conduce a la formación de primordios de nódulos que luego se diferencian en nódulos radiculares fijadores de nitrógeno que proporcionan condiciones microaeróbicas en la zona central para el funcionamiento de la enzima nitrogenasa sensible al oxígeno en los bacteroides. Dependiendo de la naturaleza transitoria y persistente de la proliferación de células huésped, los nódulos pueden ser de tipo determinado o indeterminado (Terpolilli et al., 2012; Kondorosi et al., 2013). Los nódulos determinados no tienen meristemo y contienen una población homogénea de células simbióticas. Se desarrollan nódulos determinados, por ejemplo, en las raíces de Phaseolus vulgaris y Lotus japonicus.
por el contrario, la división celular activa se mantiene en los nódulos indeterminados. Un meristemo de nódulo está presente en la región apical (zona I) que, mediante la generación constante de nuevas células, provoca un crecimiento continuo y una forma de nódulo alargada. Las células que salen del meristemo ya no se dividen y entran en una fase de diferenciación. El hilo de infección libera las bacterias en las células sumergibles, que se diferencian gradualmente a lo largo de las 12-15 capas celulares de la zona de infección (zona II), lo que lleva al desarrollo de células simbióticas fijadoras de nitrógeno en la zona de nódulos III (Figura 1; Franssen et al., 1992). Medicago sativa, M. truncatula, Vicia sativa y Pisum sativum son ejemplos de plantas que forman nódulos indeterminados.
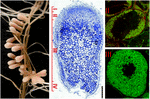
la FIGURA 1. Estructura de nódulos radiculares fijadores de nitrógeno formados en simbiosis de truncátula de S. meliloti-M. Las diferentes zonas de nódulos se indican en la sección longitudinal del nódulo: I) meristemo, II) zona de infección, III) zona de fijación de nitrógeno, IV) zona de senescencia. Las células simbióticas de la zona II contienen los endosimbiontes diferenciadores, mientras que en la zona III el citoplasma del huésped está lleno de bacteroides largos fijadores de nitrógeno. Los endosimbiontes teñidos con Syto9 tienen fluorescencia verde.
El crecimiento de Células Simbióticas Implica la Amplificación del Genoma del Huésped mediante Ciclos de Endoreduplicación
Se puede observar un aumento extremo de las células vegetales tanto en los nódulos determinados como en los indeterminados. El citoplasma de una célula simbiótica fijadora de nitrógeno alberga alrededor de 50.000 bacteroides. Para acomodar un número tan alto de endosimbiontes, las células huésped crecen. En los nódulos de M. truncatula, el volumen de las células fijadoras de nitrógeno es 80 veces mayor que el de las células meristemáticas diploides. El crecimiento de células infectadas se produce paso a paso en la zona II y es la consecuencia de endoreduplicación (RE) repetida del genoma sin mitosis. En la zona II, la maquinaria del ciclo celular sigue activa, pero la falta de ciclinas mitóticas inhibe la mitosis y transforma los ciclos mitóticos en ciclos de endoreduplicación(Cebolla et al., 1999). Esto se logra mediante la proteína CCS52A de interruptor de ciclo celular que, mediante la destrucción de las ciclinas mitóticas, induce rondas repetidas de duplicación del genoma que conducen a la formación de células poliploides de crecimiento gradual (Roudier et al., 2003; Kondorosi y Kondorosi, 2004). En las especies de Medicago, los niveles de ploidía pueden alcanzar 64C, lo que representa un contenido de ADN 64 veces mayor en comparación con las células haploides (C corresponde al contenido de ADN haploide; Vinardell et al., 2003). La regulación a la baja de CCS52A en M. truncatula no tuvo efecto en la formación de primordios, pero fue perjudicial para la diferenciación de nódulos, lo que indica que los ciclos de RE y la formación de células grandes altamente poliploides son esenciales para el funcionamiento de los nódulos (Vinardell et al., 2003). Curiosamente, las células corticales que contienen hongos AM también son poliploides, así como las células gigantes de la raíz que se alimentan de nematodos (Favery et al., 2002; Genre et al., 2008). De manera similar, las células simbióticas de insectos, los bacteriocitos que albergan endosimbiontes intracelulares, también son grandes y poliploides (Nakabachi et al., 2010). En las plantas de angiospermas, la poliploidía es frecuente y el patrón hereditario específico de poliploidía en diferentes órganos, tejidos y tipos de células sugiere que podría ser una fuente importante de la fisiología especializada de las células huésped (Nagl, 1976; Edgar et al., 2014). Además del crecimiento celular, las múltiples copias de genes, la falta de condensación cromosómica puede contribuir a una mayor actividad transcripcional y metabólica. Sin embargo, la asociación de la poliploidía con diferentes funciones celulares sugiere un impacto de la poliploidía también en la arquitectura de los nucleosomas y en el epigenoma que controla la activación o represión de regiones genómicas específicas. En consecuencia, el contenido del genoma poliploide de las células simbióticas parece ser un requisito previo para la diferenciación de nódulos y para la expresión de la mayoría de los genes del huésped simbiótico(Maunoury et al., 2010).
Diferentes Destinos de Bacteroides Fijadores de Nitrógeno
Las bacterias liberadas de la TI están presentes en el citoplasma del huésped como estructuras en forma de orgánulos, llamadas simbiosomas. Las bacterias no tienen contacto directo con el citoplasma, ya que están rodeadas por una membrana peribacteroide, conocida también como membrana simbiosómica (SM). El bacteroide, el SM y el espacio entre ellos forman el simbiosoma (Catalano et al., 2004). El SM durante su formación refleja su origen en la membrana plasmática, las modificaciones posteriores de su composición abren nuevas funciones especializadas en la interfaz huésped-endosimbionte(Limpens et al., 2009; Ivanov et al., 2012; Brear et al., 2013; Sinharoy et al., 2013). Los bacteroides se multiplican en las células del nódulo del huésped en crecimiento hasta una cierta densidad celular, se adaptan al estilo de vida endosimbiótico y a las condiciones microaeróbicas y maduran a bacteroides fijadores de nitrógeno. Sin embargo, la forma y la fisiología de los bacteroides pueden ser sorprendentemente diferentes en las distintas legumbres. En ciertos hospedantes de leguminosas, los bacteroides fijadores de nitrógeno tienen la misma morfología que las células cultivadas; este tipo de bacteroides puede revertir a la forma de vida libre. En otras asociaciones, los bacteroides se transforman irreversiblemente en endosimbiontes poliploides, agrandados y no cultivables. Estos bacteroides diferenciados terminales pueden ser alargados e incluso ramificados y de 5 a 10 veces más largos que las células de vida libre o pueden ser esféricos de 8 a al menos 20 veces el genoma amplificado dependiendo del huésped (Mergaert et al., 2006; Nakabachi et al., 2010). La diferenciación terminal de bacteroides está controlada por el huésped, evolucionada en múltiples ramas de la familia de las Leguminosas, lo que indica una ventaja para el huésped y un rendimiento simbiótico probablemente mayor (Oono et al., 2010). La diferenciación bacteroide terminal es la mejor dilucidada en la simbiosis truncatula de S. meliloti – M. En M. nódulos de truncátula, los eventos más visibles de diferenciación bacteroide terminal ocurren en la zona II. La multiplicación de bacteroides se detiene en el centro de la zona II, donde comienza la elongación celular y la amplificación uniforme de los múltiples replicones mediante ciclos de endoreduplicación. A lo largo de 2-3 capas celulares en el borde de la zona II y III (llamada interzona) es visible un crecimiento repentino de bacteroides que alcanza prácticamente su tamaño final, sin embargo, la fijación de nitrógeno solo tiene lugar en la zona III.
Los péptidos Huésped Gobiernan la diferenciación bacteroide
La comparación de transcriptomas de nódulos de legumbres con diferenciación bacteroide reversible e irreversible reveló la existencia de varios cientos de pequeños genes que solo estaban presentes en el genoma de aquellas plantas huésped donde la diferenciación bacteroide era terminal. En M. truncatula, las células nodulares producen al menos 600 péptidos simbióticos específicos de nódulos (symPEPs). Los genes symPEP solo se activan en las células simbióticas poliploides infectadas por S. meliloti (Kevei et al., 2002; Mergaert et al., 2003), sin embargo, algunos conjuntos al principio, otros durante las etapas posteriores del desarrollo de nódulos. Una gran porción, más de 500 genes codifican péptidos ricos en cisteína específicos de nódulos (NCR) (Mergaert et al., 2003; Alunni et al., 2007; Nallu et al., 2014). Los péptidos NCR están dirigidos a los bacteroides y cuando se bloqueó su entrega a los endosimbiontes, se abolió la diferenciación bacteroide, demostrando que los péptidos son responsables de la diferenciación terminal de los bacteroides de S. meliloti (Van de Velde et al., 2010). La alta variedad de secuencias y los patrones de expresión característicos de los genes NCR sugieren diversidad en sus funciones, modos de acción y dianas bacterianas en diferentes etapas de maduración bacteroide (Figura 2). Sin embargo, ¿por qué la célula huésped produce un arsenal de NCR? ¿Cuál puede ser la ventaja de un repertorio de péptidos tan diverso? ¿Es necesario para la interacción del huésped con varias bacterias? Los socios simbióticos de M. truncatula son S. meliloti y S. medicae, sin embargo, en el suelo hay innumerables variantes de cepas de ambas especies. M. el truncátula también está representado por muchos ecotipos y accesiones diferentes que difieren en el número, las secuencias y el perfil de expresión de los genes NCR y en sus interacciones simbióticas con diferentes cepas de S. meliloti y S. medicae (Nallu et al., 2014; Roux et al., 2014). Mientras que un nódulo contiene un solo tipo de bacteria, los diferentes nódulos en el mismo sistema radicular pueden poseer poblaciones bacterianas distintas. Es posible que la planta que reconoce los diversos endosimbiontes los manipule con un repertorio de péptidos específico de la cepa. Estas diferencias pueden agregar un nivel de control adicional para la especificidad del huésped-simbionte y, por lo tanto, para la eficiencia de la nodulación.

la FIGURA 2. Expresión diferencial de genes simPEP en nódulos de M. truncatula. Señal negra: hibridación in situ, señal azul: actividad de GUS de fusiones promotor-GUS de symPEP en nódulos transgénicos.
Aunque los SIMPEP representan clases de péptidos únicas, sus estructuras se asemejan a los péptidos antimicrobianos (AMPs). Los amplificadores con un amplio espectro de actividad de destrucción de células microbianas son con mayor frecuencia provocadores catiónicos de muerte celular por formación de poros, alteración de membranas y consecuente lisis de células microbianas. El hecho de que la capacidad de división celular se pierda definitivamente durante la diferenciación endosimbionte indica que al menos ciertos simPEPs tienen actividades antimicrobianas. De hecho, el tratamiento de bacterias con RNC catiónicos sintéticos provocó la eliminación rápida y eficiente, dependiente de la dosis, de varias bacterias Gram negativas y Gram positivas, incluidos importantes patógenos humanos y vegetales (Van de Velde et al., 2010; Tiricz et al., 2013). Este efecto destructor ex-planta se correlacionó con la permeabilización de las membranas microbianas, sin embargo, los SIMPEP en su entorno natural, en las células de nódulos, no permeabilizan las membranas bacterianas y no matan a los endosimbiontes. Lo más probable es que las concentraciones de péptidos en los nódulos sean significativamente más bajas que las aplicadas en los ensayos in vitro. Además, los péptidos catiónicos se producen junto con péptidos aniónicos y neutros en la misma célula, y la posible combinación de unas pocas decenas o cientos de péptidos con varias cargas e hidrofobicidad podría neutralizar el efecto bactericida directo de los péptidos catiónicos.
La participación de amperios o péptidos similares a amperios no es única para la simbiosis Rizobio-leguminosa. En el picudo Sitophilus, las células simbióticas producen el péptido antimicrobiano coleoptericina – A (ColA) que provoca el desarrollo de endosimbiontes filamentosos gigantes inhibiendo la división celular y protege los tejidos de insectos vecinos de la invasión bacteriana (Login et al., 2011). En este sistema, un solo péptido es suficiente para la diferenciación de los endosimbiontes obligados transmitidos verticalmente, a diferencia de los nódulos que operan con cientos de simPEPs y pueden albergar innumerables variantes de deformación como sus endosimbiontes. En la simbiosis pulgón-Buchnera, las células huésped también producen péptidos específicos de bacteriocitos, incluidos los péptidos ricos en cisteína (BCR) que se asemejan a los péptidos Medicago NCR, sin embargo, aún no se han reportado las funciones de estos péptidos simbióticos (Shigenobu y Stern, 2013).
NCR247: Un ejemplo para el análisis del transcriptoma del Efector de Host Multiobjetivo
de M. nódulos de truncátula en diferentes etapas de su desarrollo, microdisección láser de regiones de nódulos, hibridación in situ, inmunolocalización de péptidos seleccionados y fusiones génicas de promotor-reportero de simPEP en nódulos transgénicos permiten mapear la acción de péptidos individuales en las células simbióticas desde la infección temprana hasta el estado de fijación tardía de nitrógeno. NCR247 se expresa en las capas celulares más antiguas de la zona II y en la interzona donde se detiene la división celular bacteriana y se produce un notable alargamiento de los endosimbiontes (Farkas et al., 2014). Este pequeño péptido catiónico mató eficazmente a varios microbios in vitro y el análisis in silico indicó sus capacidades extremas de unión a proteínas. NCR247 marcado con FITC entró en el citosol bacteriano donde sus interacciones con numerosas proteínas bacterianas eran posibles. Los socios de unión se identificaron mediante el tratamiento de bacterias o bacteroides de S. meliloti con péptidos estreptocócicos/marcados con BANDERAS, seguido de cromatografía de afinidad e identificación de socios interactuantes con LC-MS/MS y análisis occidental (Farkas et al., 2014).
Uno de los interactores fue la proteína de división celular FtsZ, que desempeña un papel fundamental en la división celular. Se sabe que una serie de péptidos antibióticos ejercen un efecto bactericida o bacteriostático a través de la interacción con FtsZ, inhibiendo su polimerización, lo que dificulta la formación adecuada del anillo Z y el tabique (Handler et al., 2008). NCR247 se co-purificó con FtsZ a partir del citoplasma bacteriano y se demostró que interrumpía la formación del tabique. NCR035 que muestra in vitro también efecto bactericida y producido en las mismas células simbióticas que NCR247 se acumula en el tabique de división, lo que indica la acción simultánea o consecutiva de estos péptidos y la evolución de estrategias de múltiples huéspedes para inhibir la proliferación endosimbionte. Otro estudio mostró que la expresión de genes importantes de división celular, incluidos los genes necesarios para la función del anillo Z, se atenuaron fuertemente en las células tratadas con NCR247 (Penterman et al., 2014). El pretratamiento de bacterias con concentraciones sub-letales de NCR247 abolió la localización de FITC-NCR035 al tabique y provocó elongación celular (Farkas et al., 2014).
Las proteínas ribosómicas fueron las parejas interactuantes NCR247 más abundantes. Se observó que la NCR247 inhibe fuertemente la síntesis de proteínas bacterianas de forma dependiente de la dosis tanto in vivo como in vitro (Farkas et al., 2014). Estos resultados sugirieron que un modo de la acción del péptido NCR247 se une a los ribosomas tanto en las células bacterianas como en los bacteroides. Curiosamente, se observó un patrón alterado y una complejidad reducida de las proteínas que interactúan en los bacteroides. En consecuencia, el nivel de expresión general de las proteínas ribosómicas fue en promedio 20 veces menor en los bacteroides que en las células de vida libre con diferente abundancia relativa de transcripciones de proteínas ribosómicas individuales. La diversificación de ribosomas en bacteroides puede tener un papel significativo al contribuir a la traducción avanzada de proteínas específicas, apoyando así la fisiología especializada y exigente en energía de la función de fijación de nitrógeno altamente abundante.
El acompañante GroEL también fue un compañero de interacción directa de NCR247 (Farkas et al., 2014). De las 5 proteínas GroEL, GroEL1 o GroEL2 son suficientes para la supervivencia, mientras que GroEL1 expresado a alto nivel en el nódulo es esencial para la simbiosis (Bittner et al., 2007). Es necesario para la activación completa de los genes de nodulación y el ensamblaje del complejo nitrogenasa. GroEL posee una versatilidad funcional extrema al interactuar con cientos de proteínas. La interacción NCR247-GroEL1 puede tener consecuencias directamente sobre GroEL, pero también indirectamente sobre los sustratos GroEL y los procesos biológicos asociados. Ausencia de infección bacteriana gravemente afectada por GroEL1 y mantenimiento y diferenciación de bacteroides, lo que demuestra la necesidad general de GroEL1 en todas las etapas del desarrollo de nódulos fijadores de nitrógeno.
La participación de los péptidos GroEL y huésped en las interacciones microbio-huésped no es única para la simbiosis rizobio-leguminosa. En las células simbióticas del gorgojo, la coleoptericina A (ColA) interactúa también con GroEL (Login et al., 2011). GroEL también juega un papel importante en el mantenimiento de endosimbiontes (Moran, 1996; Kupper et al., 2014). Como la mayoría de los sistemas simbióticos están aún inexplorados y las herramientas genómicas y proteómicas de alto rendimiento solo están disponibles recientemente, solo podemos predecir que la diferenciación endosimbionte mediada por péptidos del huésped, así como la amplificación del genoma de las células huésped y los endosimbiontes diferenciados terminales son estrategias generales de simbiosis.
Conclusión
Las bacterias simbióticas y patógenas utilizan enfoques similares para interactuar con sus huéspedes y sobrevivir dentro de las células huésped, incluso si los resultados de estas interacciones son sorprendentemente diferentes. Las plantas y los animales pueden generar respuestas inmunitarias innatas a los microorganismos a partir de la percepción de mAmps (patrones moleculares asociados a microorganismos). Esta percepción resulta en la activación de cascadas de señalización y la producción de efectores antimicrobianos. Péptidos huésped tipo AMP, como el M. los péptidos truncátula NCR o el péptido antimicrobiano gorgojo de cola desempeñan un papel fundamental y multifacético en el control de la multiplicación y diferenciación de endosimbiontes, restringiendo así la presencia de bacterias a las células simbióticas. Por lo tanto, los organismos huéspedes utilizan estos péptidos efectores para domesticar e incluso contratar invasores microbianos seleccionados para el servicio.
Declaración de Conflicto de Intereses
Los autores declaran que la investigación se realizó en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Reconocimientos
El trabajo en nuestros laboratorios está respaldado por la Beca Avanzada»SYM-BIOTICS» del Consejo Europeo de Investigación a Éva Kondorosi (beca número 269067) y por TÁMOP-4.2.2.A-11/1 / KONV-2012-0035 financiado por la Unión Europea y cofinanciado por el Fondo Social Europeo.
Abdel-Lateif, K., Bogusz, D., and Hocher, V. (2012). El papel de los flavonoides en el establecimiento de endosimbiosis de raíces de plantas con hongos micorrizas arbusculares, rizobias y bacterias Frankia. Señal de Planta. Behav. 7, 636–641. doi: 10.4161 / psb.20039
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Alunni, B., Kevei, Z., Redondo-Nieto, M., Kondorosi, A., Mergaert, P., and Kondorosi, E. (2007). Genomic organization and evolutionary insights on GRP and NCR genes, two large nodule-specific gene families in Medicago truncatula. Mol. Plant Microbe Interact. 20, 1138–1148. doi: 10.1094/MPMI-20-9-1138
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Barnett, M. J., Fisher, R. F., Jones, T., Komp, C., Abola, A. P., Barloy-Hubler, F., et al. (2001). Secuencia de nucleótidos y funciones predichas de todo el megaplásmido Sinorhizobium meliloti pSymA. Proc. Natl. Acad. Sci. U. S. A. 98, 9883-9888. doi: 10.1073 / pnas.161294798
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Bittner, A. N., Foltz, A., and Oke, V. (2007). Solo se requiere uno de los cinco genes GroEL para la viabilidad y la simbiosis exitosa en Sinorhizobium meliloti. J. Bacteriol. 189, 1884–1889. doi: 10.1128 / JB.01542-06
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Brear, E. M., Day, D. A., and Smith, P. M. (2013). Iron: an essential micronutrient for the legume-Rhizobium symbiosis. Front. Plant Sci. 4:359. doi: 10.3389/fpls.2013.00359
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Catalano, C. M., Lane, W. S., and Sherrier, D. J. (2004). Biochemical characterization of symbiosome membrane proteins from Medicago truncatula root nodules. Electrophoresis 25, 519–531. doi: 10.1002/elps.200305711
Resumen de Pubmed / Texto Completo de Pubmed / Texto completo cruzado
Capela, D., Barloy-Hubler, F., Gouzy, J., Bothe, G., Ampe, F., Batut,J., et al. (2001). Análisis de la secuencia cromosómica del simbionte de leguminosas Sinorhizobium meliloti cepa 1021. Proc. Natl. Acad. Sci. U. S. A. 98, 9877-9882. doi: 10.1073 / pnas.161294398
Resumen de Pubmed / Texto Completo de Pubmed/Texto completo cruzado
Cebolla, A., Vinardell, J. M., Kiss, E., Oláh, B., Roudier, F., Kondorosi,A., et al. (1999). El inhibidor mitótico ccs52 es necesario para endoreduplicación y agrandamiento celular dependiente de ploidía en plantas. EMBO J. 18, 4476-4484. doi: 10.1093 / emboj / 18.16.4476
Texto completo cruzado
Chen, W. M., Moulin, L., Bontemps, C., Vandamme, P., Béna, G., and Boivin-Masson, C. (2003). La fijación simbiótica de nitrógeno de leguminosas por β-proteobacterias está muy extendida en la naturaleza. J. Bacteriol. 185, 7266–7272. doi: 10.1128 / JB.185.24.7266-7272.2003
CrossRef Texto Completo
Edgar, B. A., Zielke, N. y Gutiérrez, C. (2014). Endociclos: una innovación evolutiva recurrente para el crecimiento celular postmitótico. NAT. Reverendo Mol. Celular. Biol. 15, 197–210. doi: 10.1038 / nrm3756
Pubmed Resumen / Texto completo de Pubmed/Texto cruzado
Farkas, A., Maróti, G., Dürgõ, H., Györgypál, Z., Lima, R. M., Medzihradszky,K. F., et al. (2014). El péptido simbiótico NCR247 de Medicago truncatula contribuye a la diferenciación bacteroide a través de múltiples mecanismos. Proc. Natl. Acad. Sci. U. S. A. 111, 5183-5188. doi: 10.1073 / pnas.1404169111
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Favery, B., Complainville, A., Vinardell, J. M., Lecomte, P., Vaubert, D., Mergaert, P.,et al. (2002). The endosymbiosis-induced genes ENOD40 and CCS52a are involved in endoparasitic-nematode interactions in Medicago truncatula. Mol. Plant-Microbe Interact. 15, 1008–1013. doi: 10.1094/MPMI.2002.15.10.1008
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Franssen, H. J., Vijn, I., Yang, W. C., and Bisseling, T. (1992). Developmental aspects of the Rhizobium-legume symbiosis. Plant Mol. Biol. 19, 89–107. doi: 10.1007
CrossRef Full Text
Fraysse, N., Couderc, F., and Poinsot, V. (2003). Surface polysaccharide involvement in establishing the Rhizobium–legume symbiosis. Eur. J. Biochem. 270, 1365–1380. doi: 10.1046/j.1432-1033.2003.03492.x
CrossRef Full Text
Genre, A., Chabaud, M., Faccio, A., Barker, D. G., and Bonfante, P. (2008). El ensamblaje del aparato de prepenetración precede y predice los patrones de colonización de hongos micorrícicos arbusculares dentro de la corteza radicular de Medicago truncatula y Daucus carota. Célula Vegetal 20, 1407-1420. doi: 10.1105 / tpc.108.059014
Resumen de Pubmed | Texto completo de Pubmed | Texto completo cruzado
Handler, A. A., Lim, J. E., and Losick, R. (2008). Inhibidor peptídico de la citocinesis durante la esporulación en Bacillus subtilis. Mol. Microbiol. 68, 588–599. doi: 10.1111 / j. 1365-2958. 2008. 06173.x
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Ivanov, S., Fedorova, E. E., Limpens, E., De Mita, S., Genre, A., Bonfante, P.,et al. (2012). Rhizobium–legume symbiosis shares an exocytotic pathway required for arbuscule formation. Proc. Natl. Acad. Sci. U.S.A. 109, 8316–8321. doi: 10.1073/pnas.1200407109
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Kevei, Z., Vinardell, J. M., Kiss, G. B., Kondorosi, A., and Kondorosi, E. (2002). Las proteínas ricas en glicina codificadas por una familia de genes específicos de nódulos están implicadas en diferentes etapas del desarrollo de nódulos simbióticos en Medicago spp. Mol. Los Microbios de las Plantas Interactúan. 15, 922–931. doi: 10.1094 / MPMI.2002.15.9.922
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Kondorosi, E., and Kondorosi, A. (2004). Endoreduplicación y activación del complejo promotor de anafases durante el desarrollo de células simbióticas. FEBS Lett. 567, 152–157. doi: 10.1016 / j. febslet.2004.04.075
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Kondorosi, E., Mergaert, P., and Kereszt, A. (2013). Un paradigma para la vida endosimbiótica: diferenciación celular de la bacteria Rhizobium provocada por factores de la planta huésped. Annu. Apo. Microbiol. 67, 611–628. doi: 10.1146 / annurev-micro-092412-155630
Texto completo cruzado
Kupper, M., Gupta, S. K., Feldhaar, H., and Gross, R. (2014). Funciones versátiles del GroEL de chaperonina en las interacciones entre microorganismos e insectos. FEMS Microbiol. Lett. 353, 1–10. doi: 10.1111/1574-6968.12390
Pubmed Abstract | Pubmed Full Text/CrossRef Full Text
Limpens, E., Ivanov, S., van Esse, W., Voets, G., Fedorova, E., and Bisseling, T. (2009). Los simbiosomas fijadores de N2 de Medicago adquieren el marcador de identidad endocítico Rab7, pero retrasan la adquisición de la identidad vacuolar. Célula Vegetal 21, 2811-2828. doi: 10.1105 / tpc.108.064410
Resumen de Pubmed / Texto Completo de Pubmed/Texto completo cruzado
Login, F. H., Balmand, S., Vallier, A., Vincent-Monégat, C., Vigneron, A., Weiss-Gayet, M., et al. (2011). Los péptidos antimicrobianos mantienen los endosimbiontes de insectos bajo control. Science 334, 362-365. doi: 10.1126 / ciencia.1209728
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
MacLean, A. M., Finan, T. M., and Sadowsky, M. J. (2007). Genomas de las bacterias fijadoras de nitrógeno simbióticas de las legumbres. Fisiol Vegetal. 144, 615–622. doi: 10.1104/p 107.101634
Pubmed Abstract | Pubmed Texto Completo | CrossRef Texto Completo
Maunoury, N., Redondo-Nieto, M., Bourcy, M., Van de Velde, W., Alunni, B., Laporte, P., et al. (2010). La diferenciación de células simbióticas y endosimbiontes se acopla a dos transcriptomas-interruptores en la nodulación del tronco de Medicago. PLoS ONE 5: e9519. doi: 10.1371 / journal.ponga.0009519
Resumen de Pubmed / Texto Completo de Pubmed/Texto cruzado
Mergaert, P., Nikovics, K., Kelemen, Z., Maunoury, N., Vaubert, D., Kondorosi, A., et al. (2003). Una familia novedosa en Medicago truncatula que consta de más de 300 genes específicos de nódulos que codifican pequeños polipéptidos secretados con motivos de cisteína conservados. Fisiol Vegetal. 132, 161–173. doi: 10.1104/pp.102.018192
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Mergaert, P., Uchiumi, T., Alunni, B., Evanno, G., Cheron, A., Catrice, O.,et al. (2006). Eukaryotic control on bacterial cell cycle and differentiation in the Rhizobium-legume symbiosis. Proc. Natl. Acad. Sci. U.S.A. 103, 5230–5235. doi: 10.1073/pnas.0600912103
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Moran, N. A. (1996). Evolución acelerada y trinquete de Muller en bacterias endosimbióticas. Proc. Natl. Acad. Sci. U. S. A. 93, 2873-2878. doi: 10.1073 / pnas.93.7.2873
Texto completo cruzado
Moran, N. A., McCutcheon, J. P., and Nakabachi, A. (2008). Genómica y evolución de simbiontes bacterianos heredables. Annu. Reverendo Genet. 42, 165–690. doi: 10.1146 / annurev.genet.41.110306.130119
Texto completo cruzado
Nakabachi, A., Koshikawa, S., Miura, T., and Miyagishima, S. (2010). Tamaño del genoma de Pachypsylla venusta (Hemiptera: Psyllidae) y la ploidía de su bacteriocito, la célula huésped simbiótica que alberga bacterias mutualistas intracelulares con el genoma celular más pequeño. Toro. Entomol. Res. 100, 27 a 33. doi: 10.1017 / S0007485309006737
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nagl, W. (1976). Endoreduplicación y politenía del ADN entendida como estrategias evolutivas. Nature 261, 614-615. doi: 10.1038 / 261614a0
Pubmed Resumen / Texto completo de Pubmed / Texto cruzado
Nallu, S., Silverstein, K. A., Zhou, P., Young, N. D., and Vandenbosch, K. A. (2014). Patterns of divergence of a large family of nodule cysteine-rich peptides in accessions of Medicago truncatula. Plant J. 78, 697-705. doi: 10.1111 / tpj.12506
Resumen de Pubmed / Texto completo de Pubmed / Texto completo cruzado
Oldroyd, G. E. (2013). Habla, amigo y entra: sistemas de señalización que promueven asociaciones simbióticas beneficiosas en las plantas. NAT. Apo. Microbiol. 11, 252–263. doi: 10.1038/nrmicro2990
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Oono, R., Schmitt, I., Sprent, J. I., and Denison, R. F. (2010). Multiple evolutionary origins of legume traits leading to extreme rhizobial differentiation. New Phytol. 187, 508–520. doi: 10.1111/j.1469-8137.2010.03261.x
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Penterman, J., Abo, R. P., De Nisco, N. J., Arnold, M. F., Longhi, R., Zanda, M.,et al. (2014). Los péptidos de la planta huésped provocan una respuesta transcripcional para controlar el ciclo celular de Sinorhizobium meliloti durante la simbiosis. Proc. Natl. Acad. Sci. U. S. A. 111, 3561-3566. doi: 10.1073 / pnas.1400450111
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Price, D. R., Duncan, R. P., Shigenobu, S., and Wilson, A.C. (2011). Expansión del genoma y expresión diferencial de transportadores de aminoácidos en la interfaz simbiótica pulgón/buchnera. Mol. Biol. Evol. 28, 3113–3126. doi: 10.1093/molbev/msr140
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Roudier, F., Fedorova, E., Lebris, M., Lecomte, P., Györgyey, J., Abad, P.,et al. (2003). The Medicago species A2-type cyclin is auxin regulated and involved in meristem formation but dispensable for endoreduplication-associated developmental programs. Plant Physiol. 131, 1091–1103. doi: 10.1104/pp.102.011122
CrossRef Full Text
Roux, B., Rodde, N., Jardinaud, M. F., Timmers, T., Sauviac, L., Cottret, L.,et al. (2014). Análisis integrado de la expresión génica de plantas y bacterias en nódulos radiculares simbióticos mediante microdisección por captura láser acoplada a secuenciación de ARN. Plant J. 77, 817-837. doi: 10.1111 / tpj.12442
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Shigenobu, S., and Stern, D. L. (2013). Los áfidos desarrollaron nuevas proteínas secretadas para la simbiosis con endosimbiontes bacterianos. Proc. Biol. Sci. 280, 20121952. doi: 10.1098 / rspb.2012.1952
Resumen de Pubmed / Texto Completo de Pubmed / Texto completo Cruzado
Sinharoy, S., Torres-Jerez, I., Bandyopadhyay, K., Kereszt, A., Pislariu, C. I., Nakashima, J.,et al. (2013). El regulador del factor de transcripción C2H2 de la diferenciación de simbiosomas reprime la transcripción del gen de la vía secretora VAMP721a y promueve el desarrollo de simbiosomas en el truncátula de Medicago. Célula Vegetal 25, 3584-3601. doi: 10.1105 / tpc.113.114017
Pubmed Abstract | Pubmed Texto Completo | CrossRef Texto Completo
Terpolilli, J. J., Hood, G. A., and Poole, P. S. (2012). What determines the efficiency of N(2)-fixing Rhizobium-legume symbioses? Adv. Microb. Physiol. 60, 325–389. doi: 10.1016/B978-0-12-398264-3.00005-X
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Tiricz, H., Szûcs, A., Farkas, A., Pap, B., Lima, R. M., Maróti, G.,et al. (2013). Antimicrobial nodule-specific cysteine-rich peptides induce membrane depolarization associated changes in Sinorhizobium meliloti. Appl. Env. Microbiol. 79, 6737–6746. doi: 10.1128/AEM.01791-13
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Van de Velde, W., Zehirov, G., Szatmari, A., Debreczeny, M., Ishihara, H., Kevei, Z.,et al. (2010). Plant peptides govern terminal differentiation of bacteria in symbiosis. Science 327, 1122–1126. doi: 10.1126/science.1184057
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Vinardell, J. M., Fedorova, E., Cebolla, A., Kevei, Z., Horvath, G., Kelemen, Z.,et al. (2003). Se requiere endoreduplicación mediada por el activador complejo promotor de anafase CCS52A para la diferenciación de células simbióticas en nódulos de truncátula de Medicago. Célula vegetal 15, 2093-2105. doi: 10.1105 / tpc.014373
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Walker, S.A., and Downie, J. A. (2000). Entrada de Rhizobium leguminosarum bv. las viciae en los pelos radiculares requieren una especificidad mínima del factor de inclinación de cabeza, pero el crecimiento posterior del hilo de infección requiere nódulo o nódulo. Mol. Interacción Planta-Microbio.13, 54–62. doi: 10.1094 / MPMI.2000.13.7.754
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text