With one click, MAXIMIZE the chance to obtain functional proteins
Try it Now
Patent Number: WO2020024917A1
als toonaangevende leverancier van synthetisch DNA is GenScript toegewijd aan het gebruik van de nieuwste ontwikkelingen om bestaande algoritmen te updaten voor het verbeteren van genexpressie. Onze meest recente ontwikkeling is GenSmart ™ codon optimalisatie; een gratis, gebruiksvriendelijke online tool die u in staat stelt om het ontwerp van wild type of recombinante gensequenties te optimaliseren naar een hogere expressie in prokaryotische en zoogdierexpressiesystemen.
GenSmart™ codon optimalisatie maakt gebruik van een nieuw ontworpen algoritme gebaseerd op het “Population Immune Algorithm”, dat gebruik maakt van zowel population genetica en immunologie theorieën. In deze benadering, worden meer dan 200 factoren betrokken bij genuitdrukking, met inbegrip van GC-inhoud, codongebruik en inhoudsindex, RNase het verbinden plaatsen, en cis-handelt mRNA destabiliserende motieven gescreend en gevalideerd. In plaats van het toepassen van een single-factorsimulatie op het berekenen, wordt een multifactorbenadering aangewend om ervoor te zorgen dat alle belangrijke factoren in een bepaalde opeenvolging van het doelgen gewicht dragen. Dientengevolge, wordt elke genoptimalisatie volledig aangepast om de kans te maximaliseren om een functionele en actieve proteã ne te verkrijgen.
vergeleken met vergelijkbare tools is GenSmart™ Codonoptimalisatie toegankelijker en gebruiksvriendelijker. Alle parameters zijn geïntegreerd in het algoritme en vereisen geen input van u. Alles wat nodig is is uw sequentie en gewenste gastheer organisme; de rest wordt verzorgd door de optimalisatie tool. Bovendien kunnen uw geoptimaliseerde sequenties eenvoudig worden besteld via het GenSmart ™ Instant Quote systeem of worden gearchiveerd voor toekomstig gebruik.
opgewaardeerd! GenSmart ™ codon optimalisatie is toegepast op 50 gastheerorganismen nu, met inbegrip van CHO, mens, E. coli, T-cel, gist, Insect, Arabidopsis, maïs, rijst, soja, fruitvlieg, enz.!
waarom GenSmart™ Codonoptimalisatie
- toegankelijkheid: gratis, online tool; slechts één klik verwijderd van uw optimale sequentie
- uitgebreide factoranalyse: meer dan 200 factoren gescreend en gevalideerd
- Advanced Computing: gepatenteerd population immune algorithm to ensure the best output
- individuele Sequentiegebaseerd: Eenvoudig bestellen: naadloze integratie met GenSmart™ Instant Quote System
wat de gegevens zeggen
GenScript Data
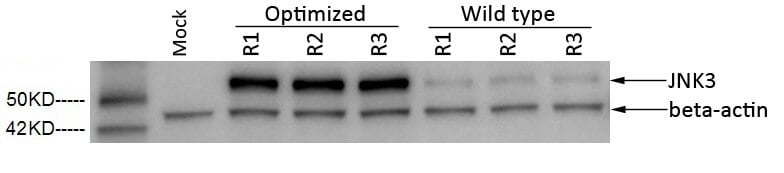
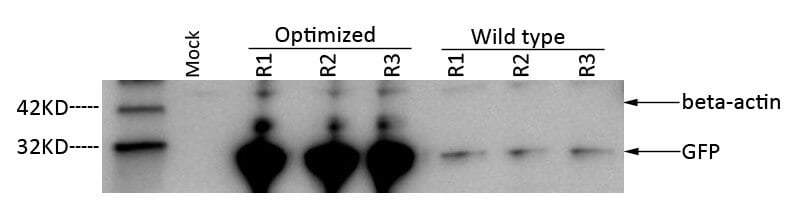
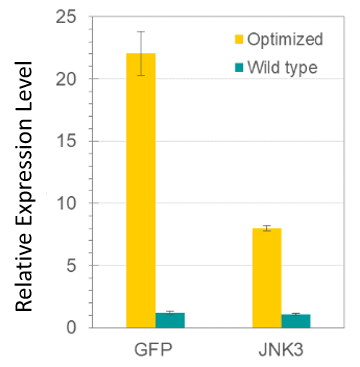
doelstelling: het beoordelen van de prestaties van GenSmart™ codon optimalisatie tool bij het verhogen van de expressie van jnk3 en GFP eiwitten.
benadering: gekloond het wild type en sequentie-geoptimaliseerde sequenties van elk gen in een expressie vector en beoordeeld eiwitexpressie van drie verschillende klonen in CHO 3E7 cellen door middel van Western blotting.
resultaten: Alle drie GFP en JNK3 klonen van opeenvolgingen die door GenSmart™ worden geoptimaliseerd toonden een significante verhoging van eiwituitdrukking door 18 en ~8 vouwen, respectievelijk, in vergelijking met niet – geoptimaliseerde inheemse genopvolgingen.
Onafhankelijke Data
Doel: Voor het beoordelen van de te bewerken efficiëntie van BE4 base-editor varianten geoptimaliseerd door verschillende codon optimalisatie tools als onderdeel van het overkoepelende doel van het onderzoeken van het effect van het gen op gen-expressie bewerken
Aanpak: Geoptimaliseerd de nucleaire lokalisatie codon van BE4 met tools van IDT, Coller, GeneArt en GenScript, en vervolgens beoordeeld genexpressie en editing efficiëntie in HEK293 cellen
resultaten: terwijl codons geoptimaliseerd door tools van GenScript (GS), GeneArt (GA) en Coller (JC) verbeterde editing efficiëntie ten opzichte van IDT ‘s, chimerische varianten geoptimaliseerd door genscript’ s codon optimalisatie alleen overtroffen andere door inducing 1,8-voudige hogere editing efficiëntie