Dans presque toutes les protéines Fe–S, les centres Fe sont tétraédriques et les ligands terminaux sont des centres thiolato-soufrés issus de résidus cystéinyliques. Les groupes sulfures sont coordonnés soit à deux, soit à trois. Trois types distincts de grappes Fe–S présentant ces caractéristiques sont les plus courants.
clustersEdit 2Fe–2S

Le système polymétallique le plus simple, le cluster, est constitué de deux ions de fer pontés par deux ions sulfures et coordonnés par quatre ligands cystéinyliques (dans les ferrédoxines Fe2S2) ou par deux cystéines et deux histidines (dans les protéines de Rieske). Les protéines oxydées contiennent deux ions Fe3+, tandis que les protéines réduites contiennent un ion Fe3+ et un ion Fe2+. Ces espèces existent dans deux états d’oxydation, (FeIII)2 et FeIIIFeII. Le domaine de soufre du fer CDGSH est également associé aux clusters 2Fe-2S.
clustersEdit 4Fe–4S
Un motif commun présente quatre ions de fer et quatre ions de sulfure placés aux sommets d’un amas de type cubane. Les centres Fe sont généralement coordonnés par des ligands cystéinyles. Les protéines de transfert d’électrons (ferrédoxines) peuvent être subdivisées en ferrédoxines à faible potentiel (de type bactérien) et à fort potentiel (HiPIP). Les ferrédoxines à potentiel faible et élevé sont liées par le schéma redox suivant:
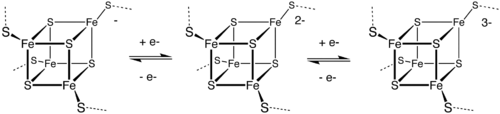
Dans HiPIP, le cluster fait la navette entre (Fe4S42+) et (Fe4S43+). Les potentiels de ce couple rédox vont de 0,4 à 0,1 V. Chez les ferrédoxines bactériennes, le couple d’états d’oxydation est (Fe4S4+) et (Fe4S42+). Les potentiels pour ce couple rédox vont de -0,3 à -0,7 V. Les deux familles d’amas 4Fe–4S partagent l’état d’oxydation Fe4S42+. La différence dans les couples redox est attribuée au degré de liaison hydrogène, qui modifie fortement la basicité des ligands de thiolate de cystéinyle. Un autre couple redox, qui est encore plus réducteur que les ferrédoxines bactériennes est impliqué dans la nitrogénase.
Certains amas 4Fe-4S se lient aux substrats et sont donc classés comme cofacteurs enzymatiques. Dans l’aconitase, le cluster Fe–S se lie à l’aconitate au seul centre Fe qui n’a pas de ligand thiolate. Le cluster ne subit pas d’oxydoréduction, mais sert de catalyseur acide de Lewis pour convertir le citrate en isocitrate. Dans les enzymes SAM radicalaires, le cluster se lie et réduit la S-adénosylméthionine pour générer un radical, qui est impliqué dans de nombreuses biosynthèses.
clustersEdit 3Fe–4S
Les protéines sont également connues pour contenir des centres, qui contiennent un fer de moins que les noyaux les plus communs. Trois ions sulfure relient deux ions fer chacun, tandis que le quatrième sulfure relie trois ions fer. Leurs états d’oxydation formels peuvent varier de + (forme all-Fe3+) à 2- (forme all-Fe2+). Dans un certain nombre de protéines fer–soufre, le cluster peut être converti de manière réversible par oxydation et perte d’un ion fer en un cluster. Par ex., la forme inactive de l’aconitase possède un et est activée par addition de Fe2+ et de réducteur.
D’autres clusters Fe–Sdit
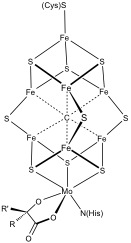
Des systèmes polymétalliques plus complexes sont courants. Les exemples incluent les clusters 8Fe et 7Fe dans la nitrogénase. Le monoxyde de carbone déshydrogénase et l’hydrogénase présentent également des clusters Fe–S inhabituels. Un cluster spécial coordonné à 6 cystéines a été trouvé dans des hydrogénases liées à la membrane tolérantes à l’oxygène.