With one click, MAXIMIZE the chance to obtain functional proteins
Try it Now
Patent Number: WO2020024917A1
In qualità di fornitore leader di DNA sintetico, GenScript è dedicato a utilizzare gli ultimi progressi per aggiornare gli algoritmi esistenti per migliorare l’espressione genica. Il nostro sviluppo più recente è GenSmart ™ Codon Optimization; uno strumento online gratuito e facile da usare che consente di ottimizzare la progettazione di sequenze geniche di tipo selvaggio o ricombinanti verso una maggiore espressione nei sistemi di espressione procarioti e mammiferi.
GenSmart™ Codon Optimization utilizza un algoritmo di nuova concezione basato sul “Population Immune Algorithm”, che sfrutta sia la genetica delle popolazioni che le teorie immunologiche. In questo approccio, più di 200 fattori coinvolti nell’espressione genica, tra cui il contenuto di GC, l’uso di codoni e l’indice di contenuto, i siti di splicing della RNasi e i motivi destabilizzanti dell’mRNA ad azione cis vengono esaminati e convalidati. Invece di applicare una simulazione a fattore singolo al calcolo, viene impiegato un approccio multifattoriale per garantire che tutti i fattori chiave in una determinata sequenza genica bersaglio portino peso. Di conseguenza, ogni ottimizzazione genica è completamente personalizzata per massimizzare la possibilità di ottenere una proteina funzionale e attiva.
Rispetto a strumenti simili, GenSmart ™ Codon Optimization è più accessibile e facile da usare. Tutti i parametri sono integrati nell’algoritmo e non richiedono alcun input da parte tua. Tutto ciò che è necessario è la sequenza e l’organismo ospite desiderato; il resto è curato dallo strumento di ottimizzazione. Inoltre, le sequenze ottimizzate possono essere facilmente ordinate tramite il sistema di preventivi istantanei GenSmart™ o archiviate per un uso futuro.
Aggiornato! L’ottimizzazione del codone di GenSmart™ è stata applicata a 50 organismi ospiti ora, compreso CHO, umano, E. coli, cellula T, lievito, insetto, Arabidopsis, mais, riso, soia, mosca della frutta, ecc.!
Perché GenSmart™ Codon Optimization
- Accessibilità: strumento gratuito e online; basta un clic per ottenere la sequenza ottimale
- Analisi fattoriale completa: oltre 200 fattori selezionati e convalidati
- Calcolo avanzato: algoritmo immunitario della popolazione brevettato per garantire il miglior output
- : Calcolo altamente personalizzato per evitare distorsioni di allocazione del peso su fattori chiave
- Facile ordinazione: integrazione senza soluzione di continuità con GenSmart™ Instant Quote System
Cosa dicono i dati
Dati GenScript
Obiettivo: Valutare le prestazioni dello strumento di ottimizzazione del codone GenSmart™ per aumentare l’espressione
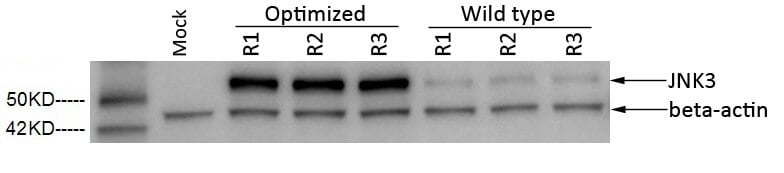
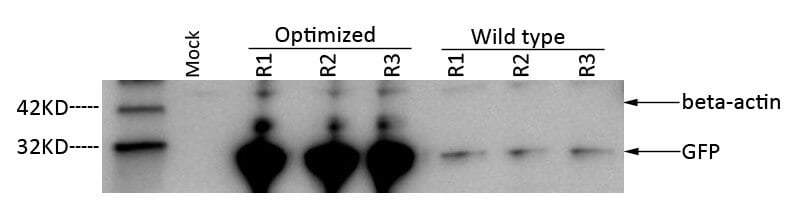
Approccio: clonato il tipo selvaggio e sequenze ottimizzate per sequenza di ciascun gene in un vettore di espressione e valutata l’espressione proteica da tre diversi cloni in cellule CHO 3E7 attraverso Western blotting.
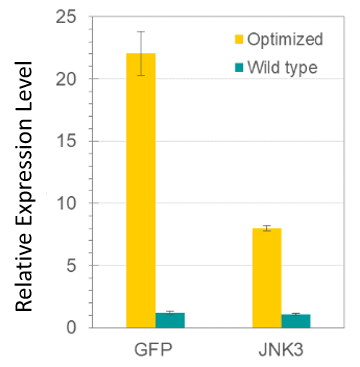
Risultati: Tutti e tre i cloni GFP e JNK3 delle sequenze ottimizzate da GenSmart™ hanno mostrato un aumento significativo dell’espressione proteica di 18 e ~8 volte, rispettivamente, rispetto alle sequenze geniche native non ottimizzate.
di Dati Indipendenti
Obiettivo: Per valutare l’editing efficienza di BE4 base editor varianti ottimizzate da diversi codone di strumenti di ottimizzazione, come parte di un obiettivo più generale di indagare l’effetto dell’espressione genica genica modifica
Approccio: Ottimizzato il codone di localizzazione nucleare di BE4 con strumenti di IDT, Coller, GeneArt e GenScript, e quindi valutato l’espressione genica e l’efficienza di editing nelle cellule HEK293
Risultati: Mentre i codoni ottimizzati dagli strumenti di GenScript (GS), GeneArt (GA) e Coller (JC) hanno migliorato l’efficienza di editing rispetto agli IDT, le varianti chimeriche ottimizzate dall’ottimizzazione del codone di GenScript hanno